جلد 21، شماره 2 - ( تابستان 1403 )
جلد 21 شماره 2 صفحات 150-138 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Masoudi M, Sharifi Z, Maghsudlu M. Molecular detection of HTLV-1 provirus from DNA released from cells in plasma, in cases of lack of access to genomic DNA of peripheral blood mononuclear cells. bloodj 2024; 21 (2) :138-150
URL: http://bloodjournal.ir/article-1-1529-fa.html
URL: http://bloodjournal.ir/article-1-1529-fa.html
مسعودی منیره، شریفی زهره، مقصودلو مهتاب. تشخیص مولکولی پروویروس HTLV-1 از DNA آزاد شده از سلولها در پلاسما، در موارد عدم دسترسی به DNA ژنومی سلولهای تک هستهای خون محیطی. فصلنامه پژوهشی خون. 1403; 21 (2) :138-150
استاد مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون
متن کامل [PDF 802 kb]
(388 دریافت)
| چکیده (HTML) (1648 مشاهده)
مقدمه
ویروس لنفوتروپیک تیپ یک انسانی (HTLV-1) ، رتروویروس اگزوژن میباشد (1). این ویروس عضو خانواده رتروویریده و جنس دلتا ویروس است (3، 2). رتروویروسها RNA ویروسهایی هستند که فرم cDNA آنها به داخل ژنوم سلول میزبان اینتگره میشود (4). تعداد افراد مبتلا به ویروس HTLV-1 در سراسر جهان حدود 20-1 میلیون نفر تخمین زده شده است که 10-5 میلیون نفر در مناطق اندمیک زندگی میکنند (7-5، 3، 1). شیوع این ویروس در مناطق اندمیک مانند ژاپن، جنوب صحرای آفریقا، منطقه کارائیب و جنوب آمریکا زیاد میباشد. همچنین این ویروس به میزان کمتر در رومانی، ملانژی-استرالیا و ایران حضور دارد (3). در حال حاضر در ایران غربالگری ویروس HTLV-1 در اهداکنندگان خون، عمدتاً در 7 استان خراسان رضوی، خراسان شمالی، خراسان جنوبی، گلستان، البرز، اردبیل و آذربایجان شرقی انجام میشود (8).
این ویروس از سه طریق مادر به فرزند، تماس جنسی و به واسطه پزشکی از فرد به فرد دیگر منتقل میشود، که خطر انتقال ویروس از راه تزریق خون را میتوان با فیلتر کردن خون و فرآیند کاهش لکوسیت قبل از تزریق خون کاهش داد (9، 5، 3). احتمال تغییرات سرمی (Seroconversion) ویروس HTLV-1 از طریق تزریق خون حدود 60%-40% گزارش شده است. بنابراین خطر انتقال ویروس از راه تزریق خون در مناطقی با شیوع بالای ویروس حائز اهمیت باشد (10).
ویروس HTLV-1 عمدتاً سلولهای لنفوسیت TCD4+، که در ایجاد پاسخهای ایمنی دخیل هستند، را درگیر میکند (11). گسترش این ویروس عمدتاً از طریق تکثیر سلولهای آلوده از راه تماس سلول به سلول میباشد (12). در شرایط آزمایشگاهی مشاهده شده است که ویروس HTLV-1 ویریونهای آزاد کمی تولید میکند و بیشتر به صورت اینتگره درون ژنوم سلول میزبان میباشد (13). در مطالعههای اخیر ذکر شده است که ژنوم ویروس HTLV-1 به واسطه حضور DNA آزاد شده از سلولها در مایعات بیولوژیک بدن (cfDNA)، قابل شناسایی است.
cfDNAشامل قطعات اسیدنوکلئیک تکرشتهای و دو رشتهای خارج سلولی میباشد که عمدتاً ناشی از نکروز و آپوپتوز سلولی است. اخیراً دیده شده است که انتشار cfDNA از طریق میکرووزیکولهایی مانند اگزوزوم نیز امکانپذیر میباشد. cfDNA در پلاسما و مایعات بیولوژیک مانند CSF قابل شناسایی هستند. اسیدهای نوکلئیک درون زاد به طور دائم در جریان خون حضور دارند و مقدار آن در شرایط پاتولوژیک تغییر میکند. مطالعهها نشان دادهاند که میتوان از cfDNA به عنوان یک نشانگر زیستی برای تشخیص، تعیین اثر درمان و پیشآگهی بیماریها استفاده کرد (17-14).
در پژوهشهای مولکولی ویروس HTLV-1 ، عمدتاً ژنوم سلولهای تک هستهای خون محیطی و بافیکوت مورد ارزیابی قرار میگیرد. از آن جایی که طول عمر سلولهای هدف چند روز میباشد و این امکان وجود دارد که در روند جمعآوری و انتقال نمونه، سلولهای هستهدار از بین بروند، جمعآوری نمونه در مطالعههای مولکولی HTLV-1 یک چالش به حساب میآید. بنابراین اگر امکان تشخیص ژنوم ویروس HTLV-1 در نمونههای سرم و پلاسما، که شرایط نگهداری طولانی مدت دارند، وجود داشته باشد، تا حدودی میتوان این چالش را برطرف نمود. هدف از این مطالعه، بررسی حضور ویروس HTLV-1 در DNA آزاد شده از سلول در پلاسما و تعیین بار ویروسی آن در مقایسه با سلولهای تک هستهای خون محیطی (PBMC) در اهداکننـدگان خـون وستـرن بـلات مثبت بود.
مواد و روشها
این پژوهش از نوع توصیفی بود و جامعه مورد مطالعه شامل 30 اهداکننده خون ایرانی بودند که به مراکز انتقال خون کشور، به منظور غربالگری ویروس HTLV-1 مراجعه داشتند. معیار ورود افراد به این مطالعه حضور آنتی بادی علیه ویروس HTLV-1/2 (نتیجه Reactive آزمایش غربالگری CLEIA) و نتیجه مثبت آزمایش تائیدی وسترن بـلات و عـدم حضـور ویروسهـای منتقلـه از راه خــون
مانندHIV ، HCV و HBV تعیین شد.
جمعآوری نمونه:
در این مطالعه از نمونههای خون کامل استفاده شد. پس از جمعآوری، نمونهها به مدت 10 دقیقه با دور RPM 3000 سانتریفوژ شدند. لایه پلاسما جدا گردیده و در فریزر 30- درجه سانتیگراد نگهداری شد. به منظور جداسازی سلولهای تکهستهای خون محیطی از فایکول (ایران، mBio) و سانتریفیوژ شیب غلظت استفاده شد. با کمک لام نئوبار تعداد سلولهای تک هستهای خون محیطی شمارش گردید. جهت استخراج DNA ژنومی تعداد یک میلیون PBMC در حجم µL 200 تهیه شد. به منظور استخراج DNA آزاد شده از سلول در پلاسما، از µL 200 پلاسما استفاده گردید. DNA پروویروس توسط کیت استخراج اسیدهای نوکلئیک ویروسی ساخت شرکت دینابایو ایران با شماره کالانما 10025K طبق دستورالعمل شرکت سازنده استخراج گردید. غلظت و خلوص DNA استخراج شده با کمک دستگاه نانودراپ (آمریکا، دنوویکس) مورد ارزیابی قرار گرفت. نمونه DNA استخراج شده از پلاسما و PBMC تا زمان آزمایش در فریزر 30- درجه سانتیگراد نگهداری شدند. مابقی جهت نگهداری طولانی مدت در فریزر 70- درجه سانتیگراد نگهداری شدند.
تائید حضور ویروس به روش Nested PCR :
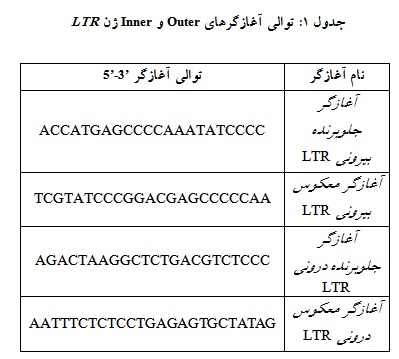
به منظور تائید حضور ویروس در نمونههای مورد استفاده، از آزمایش Nested PCR استفاده گردید. واکنش Nested PCR به منظور تکثیر ناحیه ژنی LTR ، بر روی DNA استخراج شده از پلاسما (cfDNA) و DNA ژنومی انجام شد. توالی آغازگر پیشبرنده بیرونی ناحیه ژنی LTR ، 5’ ACC ATG AGC CCC AAA TAT CCC C 3’ و توالی آغازگر معکوس بیرونی ناحیه ژنی LTR ، 5’ TCG TAT CCC GGA CGA GCC CCC AA 3’ بود. توالی آغازگر پیشبرنده درونی ناحیه ژنی LTR ، 5’ AGA CTA AGG CTC TGA CG TCT CCC 3’ و توالی آغازگر معکـوس درونـی ناحیـه ژنـی LTR ، 5’ AAT TTC TCT CCT GAG AGT GCT ATA G 3’ بود (18).
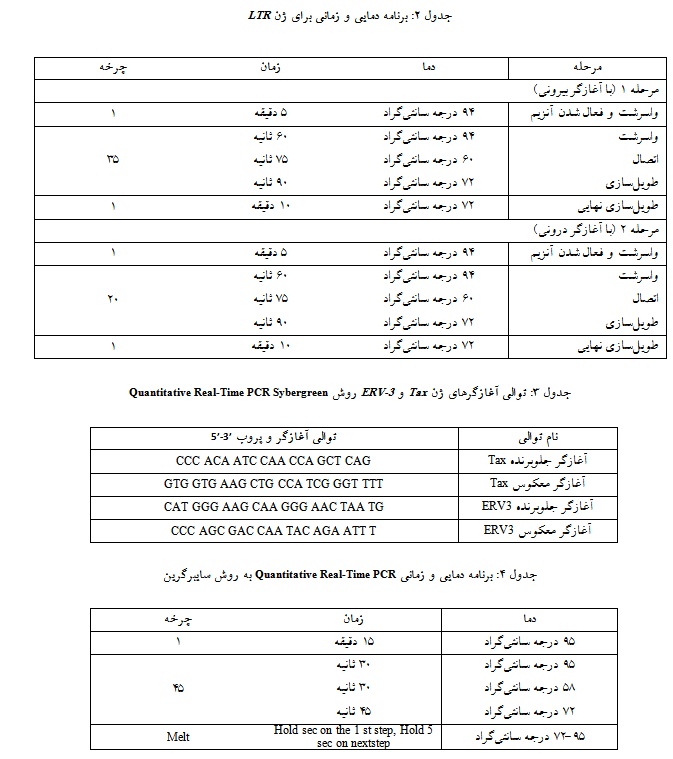
به جهت انجام واکنش Nested PCR ، µL 10 از (آلمان، آمپلیکون) X2 Master Mix ، µL 5/0 از آغازگرهای درونی و بیرونی (µL 10)، µL 4 از آب مقطر استریل و (ng 100-50) µL 5 از DNA استخراج شده از پلاسما و سلولهای تکهستهای خون محیطی استفاده شد. حجم نهایی PCR ، µL 20 تعیین شد. برنامه دمایی و زمانی واکنش Nested PCR در جدول آورده شده است (جداول 1 و 2). پس از اتمام واکنش، محصول مرحله دوم Nested PCR بر روی ژل آگارز 5/1% و رنگ DNA Green viewer در مقابل نشانگر وزن مولکولی bp 100 الکتروفورز انجام شد. سپس با دستگاه ژل داک (ایران، ATP) در مقابل نور فرا بنفش آشکارسازی گردید.
اندازهگیری بار ویروس HTLV-1 به روشQuantitative Real-Time PCR :
به منظور اندازهگیری میزان بار ویروس HTLV-1 از آزمایشQuantitative Real-Time PCR به روش سایبرگرین و ژن هدف Tax استفاده گردید. توالی آغازگر پیشبرنده ژن Tax، 5’ CCC ACA ATC CAA CCA GCT CAG 3’ و توالی آغازگر معکوس 5’ GTG GTG AAG CTG CCA TCG GGT TTT 3’ بود (19).
به جهت طبیعی کردن نتایج و کنترل داخلی، آزمایش PCR از ژن ERV3 استفاده شد. در سلولهای دیپلوئید
بحث
در این مطالعه میزان پرووایرال لود HTLV-1 در پلاسما و سلولهای تک هستهای خون محیطی اهداکنندگان خون با نتیجه وسترن بلات مثبت به روش qPCR اندازهگیری شد تا آشکار شود در مواردی که سرم یا پلاسما به جای استفاده از سلولهای تک هستهای خون محیطی برای تشخیص ویروس به کار برده میشود، تا چه حد نتایج و لود ویروس با یکدیگر مشابهت دارد. در این مطالعه، میزان میانه بار ویروس در پلاسمای ناقلین بدون علامت Copies/µL90/1 بود و میانه لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت برابر با PBMC 106/Copies 20 بود که نشان میدهد میزان لود ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از بار ویروس در Genomic DNA است. نتایج حاصل از این پژوهش، با نتایج سایر مطالعههای انجام شده در دیگر کشورها در طی سالهای اخیر مطابقت دارد و بیانکننده این موضوع میباشد که ژنوم ویروس HTLV-1 در مایعات بیولوژیک بدن مانند پلاسما قابل شناسایی میباشد. Cell-free DNA ، قطعات DNA هستند که برای اولین بار در سال 1948 در کمپلکسهای ایمنی بیماران مبتلا به لوپوس اریتروماتوز در سیستم گردش خون توصیف شدند (22). اهمیت بالینی cfDNA زمانی مشخص شد که محققان تفاوتهایی بین ویژگیهای cfDNA افراد سالم و بیمار گزارش کردند (23). توماس جوریس و همکاران در کشور انگلستان، 6 فرد سالم به عنوان گروه کنترل، 25 ناقل بدون علامت ویروس HTLV-1 ، 21 بیمار میلوپاتی ناشی از ویروس HTLV-1 (HAM) و 25 بیمار مبتلا به لوسمی سلول T بالغین (ATL) را مورد ارزیابی قرار دادند. cfDNA از پلاسمای خون و Genomic DNA از سلولهای تکهستهای خون محیطی این افراد جدا شد. به منظور تائید حضور DNA انسانی از ژن بتاگلوبین و همچنین برای اندازهگیری بار ویروس از ژن Tax و qPCR به روش سایبرگرین استفاده کردند. پروویروس HTLV-1 در 85% از cfDNA ها شناسایی شد و نشان داده شد که عدم شناسایی پروویروس در cfDNA به دلیل لود پایین ویروس در gDNA سلولهای تکهستهای خون محیطی مرتبط است. در این مطالعه بیان گردید که DNA پروویروس یکی از اجزا cfDNA پلاسما در ناقلین HTLV-1 میباشد و میزان پرووایرال لود ویروس HTLV-1 در cfDNA پلاسما تقریبا 10 برابر کمتر از پرووایرال لود ویروس در gDNA سلولهای تکهستهای خون محیطی است (24). بیماری لوسمی سلول T بزرگسالان براساس ویژگیهای بالینی و شرح حال بیمار به چهار زیر گروه بالینی Acute ، Lymphoma ، Chronic و Smoldering تقسیم میگردد. هیروکاتسویا و همکاران در کشور ژاپن cfDNA را از پلاسما و gDNA را از PBMC ، 42 ناقل بدون علامت، 9 بیمار مبتلا به Smoldering ATL ، 7 بیمار مبتلا به Chronic ATL ، 5 بیمار مبتلا به Lymphoma ATL و 4 بیمار مبتلا به Acute ATL استخراج کردند و به روش Droplet digital PCR میزان پرووایرال لود ویروس اندازهگیری شد. میانگین لود ویروس در نمونههای cfDNA پلاسما در ناقلین بدون علامت Copies/mL 7/0 ، نوع copies/mL Smoldering 5/17، نوع copies/ml Chronic ATL 3/56، نوع copies/mL Lymphoma ATL 1650 و نوع copies/mL Acute ATL 625 گزارش شد. میانگین پرووایرال لود ویروس در نمونههای gDNA سلولهای تکهستهای خون محیطی در ناقلین بدون علامت 6% ، نوع Smoldering ATL 3/16% ، نوع Chronic ATL 76% ، نوع Lymphoma ATL 5/8% و نوع Acute ATL 3/69% تعیین گردید. در این مطالعه بیان شد که بیماران مبتلا به لوسمی سلول T بالغین نوع Lymphoma بیشترین مقدار بار ویروس را در cfDNA پلاسما و کمترین میزان پرووایرال لود را در PBMC دارند (25). دانیل روزا و همکاران در کشور برزیل 48 اهداکننده خون که از نظر حضور ویروس HTLV-1 مثبت گزارش شده بودند را مورد ارزیابی قرار دادند. این افراد براساس میزان درگیری عصبی به دو گروه تقسیم شدند. گروه 1 متشکل از 23 بیمار بدون درگیری عصبی و گروه 2 متشکل از 25 بیمار با نشانههایی از درگیری عصبی بودند. در این مطالعه میزان پرووایرال لود ویروس در پلاسما و مایع مغزی نخاعی به روش Real-Time SYBR Green PCR اندازهگیری شد.
نتایج نشان دادند که میزان لود ویروس در پلاسمای افراد گروه 1 و 2 از نظر آماری تفاوت چشمگیری نداشتند. در حالی که به طور چشمگیری میزان لود ویروس در مایع مغزی ـ نخاعی گروه 2 (همراه با درگیری عصبی) نسبت به گروه 1 (بدون درگیری عصبی) بالاتر بود (03/0 p=). این نتیجه میتواند تائیدکننده این موضوع باشد که میزان لود ویروس در بیماران پاراپارزی اسپاستیک گرمسیری همراه میلوپاتی ناشی از ویروس HTLV-1 (HAM/TSP) حدود 10 تا 100 برابر بیشتر از ناقلین بدون درگیری عصبی میباشد (26). سیلوا فورتادو و همکاران در کشور برزیل میزان پرووایرال لود ویروس HTLV-1 را در 75 ناقل بدون علامت و 78 بیمار HAM مورد ارزیابی قرار دادند. در این پژوهش DNA ویروس از خون محیطی استخراج شد و با روش Real-Time SYBR Green PCR و ژن هدف Pol و آلبومین میزان لود ویروس اندازه گیری شد. در این مطالعه عنوان شده است که میزان بار ویروس در بیماران HAM (cell 104/Copies 336 cell median: 104 Copies/ 711) حدود 6 برابر بیشتر از ناقلین بدون علامت (cell 104 Copies/ 336 cell median: 104 Copies/ 117) است (27).
در این مطالعه، در 80% موارد DNA ژنومی استخراج شده از پلاسمای اهداکنندگان خون با نتیجه وسترن بلات مثبت، ناحیه ژنی LTR ویروس به روش Nested PCR شناسایی گردید. فابیو کابرال و همکاران میزان لود ویروس HTLV-1 را در DNA ژنومی آزاد شده از سلول، از پلاسمای 150 فرد مبتلا به ویروس (123 ناقل بدون علامت و 27 بیمار HAM/TSP) با هدف قرار دادن ژن Pol و کنترل داخلی آلبومین به روش TaqMan Real-Time PCR ارزیابی کردند. نتایج نشان دادند که تکثیر ژن Pol در 4% ناقل بدون علامت و 26% بیمار HAM/TSP وجود دارد (28). مطالعهها نشان دادند که خون کامل و فرآوردههای سلولی و بافتی میتوانند منجر به انتقال ویروس HTLV-1 شوند، ولی تاکنون علیرغم حضور پروویروس در cfDNA در پلاسما، انتقال ویروس HTLV-1 از فرآوردههای بدون سلول گزارش نشده است که به دلیل عدم حضور تعداد کافی ویریون HTLV-1 عفونتزا در پلاسمای بیماران میباشد. پژوهش انجام شده در این زمینه نشان داده که تعداد بسیار کمی از پروویروس های آزاد شده از سلولهای آلوده به HTLV-1 در شرایط آزمایشگاهی عفونی هستند. تخمین زده شده است که از هر 106-105 ذره تولیدی، تنها یک مورد عفونتزای باشد (30، 29).
همچنین در مطالعههایی، ژنوم ویروس در وزیکولهای خارج سلولی در پلاسمای افراد نیز شناسایی گردیده است. اخیراً ساختارهای کوچک متصل به غشا تحت عنوان وزیکولهای خارج سلولی (EVs) به دلیل حمل پروتئینها و RNA ویروس در چندین عفونت ویروسی مانند HIV-1، HTLV-1 و ویروس Zika مورد توجه قرار گرفته است. در شرایط فیزیولوژیک و پاتولوژیک تقریباً تمام سلولها EVs ترشح میکنند که میتوان آن را در مایعات بیولوژیک مانند خون، ادرار، خلط، شیر مادر، مایع مغزی- نخاعی و... شناسایی کرد. وزیکولهای خارج سلولی حاوی پروتئین، لیپید، DNA ، RNA ، محتوای ژنتیکی پاتوژنها مانند ویروسها و باکتریها و... میباشند (31). مولکولهای زیادی بر روی EVsها وجود دارد که میتوانند به تعداد زیادی از گیرندههای سلولی متصل شوند و یا از طریق فرآیند فاگوسیتوز سلولهای ایمنی محتویات وزیکولها، وارد سلولهای هدف میشوند (32، 31). مطالعههای اخیر نشان دادهاند که سلولهای آلوده به ویروس یک سری وزیکولهای خارج سلولی تولید میکنند که حاوی پروتئینها (gp61+++/Tax+++/HBZ+) و RNA ویروس میباشد. این وزیکولهای خارج سلولی قادر هستند بر روی سلولهای سالم تاثیر گذاشته و آنها را نیز درگیر کنند. همچنین در این مطالعه نشان داده شده، که میزان سنتز RNA ویروس HTLV-1 در موشهای NOG (موشهایی که زنجیره گاما رسپتور اینترلوکین 2 در آنها حذف شده است) مجاور شده با وزیکولهای خارج سلولی ویروسHTLV-1 در مقایسه با موشهای مجاور شده با وزیکولهای خارج سلولی سلولهای غیر عفونی افزایش یافته است. همچنین میزان DNA ویروس در خون، ریه، طحال، کبد و مغز موشها پس از مجاور کردن با وزیکولهای HTLV-1 اندازهگیری گردید که افزایش سطح DNA ویروس در تمام بافتها به ویژه مغز مشاهده گردید (33). مطالعهها نشان داده که وزیکولهای خارج سلولی از خون محیطی و مایع مغزی نخاعی بیماران مبتلا به HAM/TSP ، حاوی پروتئین ترانس اکتیو Tax میباشند که باعث افزایش تجمع سلولی و گسترش ویروس میشود (31). وزیکولهای خارج سلولی میتوانند باعث افزایش RNA و DNA پروویروس در خون، غدد لنفاوی و طحال شوند (34). از آن جایی که مطالعههای زیادی بر روی وزیکولهای خارج سلولی مشتق از سلولهای آلوده به HTLV-1 صورت نگرفته است و مشخص نشده است که این EVs ها عفونی هستند یا خیر، بنابراین ضروری است وزیکولهای خارج سلولی تولید شده توسط سلولهای حاوی ویروس از نظر حضور ژنوم ویروس HTLV-1، پرووایرال لود ویروس، بیان ژنهای ویروسی و عفونی بودن مورد ارزیابی قرار گیرند.
در این مطالعه میزان پرووایرال لود HTLV-1 در پلاسما و سلولهای تکهستهای خون محیطی اهداکنندگان خون با نتیجه وسترن بلات مثبت به روش qPCR سایبرگرین اندازهگیری شد تا در مواردی که سرم یا پلاسما به جای استفاده از سلولهای تک هستهای خون محیطی برای تشخیص ویروس به کار برده میشود یا لود ویروس تعیین میشود، تا چه حد نتایج با یکدیگر مشابهت دارد. در این مطالعه، میزان میانه بار ویروس در پلاسمای ناقلین بدون علامت Copies/µL90/1 بود و میانه لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت برابر با PBMC 106 Copies/ 20 بود که نشان میدهد میزان لود ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از بار ویروس در Genomic DNA است. نتایج حاصل از این پژوهش، با نتایج سایر مطالعههای انجام شده در دیگر کشورها در طی سالهای اخیر مطابقت دارد و بیانکننده این موضوع میباشد که ژنوم ویروس HTLV-1 در مایعات بیولوژیک بدن مانند پلاسما قابل شناسایی میباشد. Cell-free DNA قطعات DNA هستند که برای اولین بار در سال 1948 در کمپلکسهای ایمنی بیماران مبتلا به لوپوس اریتروماتوز در سیستم گردش خون توصیف شدند (22). اهمیت بالینی cfDNA زمانی مشخص شد که محققان تفاوتهایی بین ویژگیهای cfDNA افراد سالم و بیمار گزارش کردند (23). توماس جوریس و همکاران در کشور انگلستان، 6 فرد سالم به عنوان گروه کنترل، 25 ناقل بدون علامت ویروس HTLV-1 ، 21 بیمار مبتلا به HAM و 25 بیمار مبتلا به ATL را مورد ارزیابی قرار دادند. cfDNA از پلاسمای خون و Genomic DNA از سلولهای تکهستهای خون محیطی این افراد جدا شد. به منظور تائید حضور DNA انسانی از ژن بتاگلوبین و همچنین برای اندازهگیری بارویروس از ژن Tax و qPCR به روش سایبرگرین استفاده کردند. پروویروس HTLV-1 در 85% از cfDNA شناسایی شد و نشان داده شد که عدم شناسایی پروویروس در cfDNA به لود پایین ویروس در gDNA سلولهای تکهستهای خون محیطی مرتبط است. در این مطالعه بیان گردید که DNA پروویروس یکی از اجزای cfDNA پلاسما در ناقلین HTLV-1 میباشد و میزان پرووایرال لود ویروس HTLV-1 در cfDNA پلاسما تقریباً 10 برابر کمتر از پرووایرال لود ویروس در gDNA سلولهای تکهستهای خون محیطی میباشد (24). بیماری لوسمی سلول T بزرگسالان براساس ویژگیهای بالینی و شرح حال بیمار به چهار زیرگروه بالینی Acute ، Lymphoma، Chronic و Smoldering تقسیم میگردد. هیرو کاتسویا و همکاران در کشور ژاپن cfDNA را از پلاسما و gDNA از PBMC ، 42 ناقل بدون علامت، 9 بیمار مبتلا به Smoldering ATL، 7 بیمار مبتلا بهChronic ATL ، 5 بیمار مبتلا به Lymphoma ATL و 4 بیمار مبتلا به Acute ATL استخراج کردند و به روش Droplet digital PCR میزان پرووایرال لود ویروس اندازهگیری شد. میانگین لود ویروس در نمونههای cfDNA پلاسما در ناقلین بدون علامت copies/mL 7/0، نوع Smoldering ATL copies/mL 5/17، نوع Chronic ATL copies/mL 3/56، نوع Lymphoma ATL copies/mL 1650 و نوع Acute ATL copies/mL 625 گزارش شد. میانگین پرووایرال لود ویروس در نمونههای gDNA سلولهای تکهستهای خون محیطی در ناقلین بدون علامت 6% ، نوع Smoldering ATL 3/16%، نوع Chronic ATL 76%، نوع Lymphoma ATL 5/8% و نوع Acute ATL 3/69% تعیین گردید. در این مطالعه بیان شد که بیماران مبتلا به لوسمی سلول T بالغین نوع Lymphoma بیشترین مقدار بار ویروس را در cfDNA پلاسما و کمترین میزان پرووایرال لود را در PBMC دارند (25). دانیل رزا و همکاران در کشور برزیل 48 اهداکننده خون که از نظر حضور ویروس HTLV-1 مثبت گزارش شده بودند را مورد ارزیابی قرار دادند. این افراد براساس میزان درگیری عصبی به دو گروه تقسیم شدند. گروه 1 متشکل از 23 بیمار بدون درگیری عصبی و گروه 2 متشکل از 25 بیمار با نشانه هایی از درگیری عصبی بودند. در این مطالعه میزان پرووایرال لود ویروس در پلاسمـا و مایـع مغـزی نخاعـی بــه روش Real-Time
SYBR Green PCR اندازهگیری شد.
نتایج نشـان دادنـد کـه میـزان لود ویروس در پلاسمای
افراد گروه 1 و 2 از نظر آماری تفـاوت چشمگیـری نـداشتند. در حالی که به طور چشمگیری میزان لود ویروس در مایع مغزی نخاعی گروه 2 (همراه با درگیری عصبی) نسبت به گروه 1 (بدون درگیری عصبی) بالاتر بود (03/0 p=). این نتیجه میتواند تائیدکننده این موضوع باشد که میزان لود ویروس در بیماران HAM/TSP حدود 10 تا 100 برابر بیشتر از ناقلین بدون درگیری عصبی میباشد (26). سیلوا فورتادو و همکاران در کشور برزیل میزان پرووایرال لود ویروس HTLV-1 را در 75 ناقل بدون علامت و 78 بیمار HAM مورد ارزیابی قرار دادند. در این پژوهش DNA ویروس از خون محیطی استخراج شد و با روش Real-Time SYBR Green PCR و ژن هدف Pol و آلبومین میزان لود ویروس اندازهگیری شد. در این مطالعه عنوان شده است که میزان بار ویروس در بیماران HAM (cell 104Copies/ 336: cell median 104/Copies 711) حدود 6 برابر بیشتر از ناقلین بدون علامت (HAM 104 Copies/ 48 cell median: 104 Copies 117) است (27).
در این مطالعه، در 80% موارد DNA ژنومی استخراج شده از پلاسمای اهداکنندگان خون با نتیجه وسترن بلات مثبت ، ناحیه ژنی LTR ویروس به روش Nested PCR شناسایی گردید. فابیو کابرال و همکاران میزان لود ویروس HTLV-1 را در DNA ژنومی آزاد شده از سلول، از پلاسمای 150 فرد مبتلا به ویروس (123 ناقل بدون علامت و 27 بیمار HAM/TSP) با هدف قرار دادن ژن Pol و کنترل داخلی آلبومین به روش TaqMan Real-Time PCR ارزیابی کردند. نتایج نشان دادند که تکثیر ژن Pol در 4% ناقل بدون علامت و 26 بیمار HAM/TSP وجود دارد (28). مطالعهها نشان دادند که خون کامل و فرآوردههای سلولی و بافت میتوانند منجر به انتقال ویروس HTLV-1 شوند، ولی تاکنون علیرغم حضور پروویروس در cfDNA در پلاسما، انتقال ویروس HTLV-1 از فرآوردههای بدون سلول گزارش نشده است که به دلیل عدم حضور تعداد کافی ویریون HTLV-1 عفونت زا در پلاسمای بیماران میباشد. پژوهش انجام شده در این زمینه نشان داده که تعداد بسیار کمی از پروویروس های آزاد شده از سلولهای آلوده به HTLV-1 در شرایط آزمایشگاهی عفونی هستند. تخمین زده شده است که از هر 106-105 ذره تولیدی تنها یک مورد عفونتزا باشد (30، 29).
همچنین در مطالعههایی، ژنوم ویروس در وزیکولهای خارج سلولی در پلاسمای افراد نیز شناسایی گردیده است. اخیراً ساختارهای کوچک متصل به غشا تحت عنوان وزیکولهای خارج سلولی (EVs) به دلیل حمل پروتئینها و RNA ویروس در چندین عفونت ویروسی مانند HIV-1، HTLV-1 و ویروس Zika مورد توجه قرار گرفته است. در شرایط فیزیولوژیک و پاتولوژیک تقریبا تمام سلولها EVs ترشح میکنند که میتوان آن را در مایعات بیولوژیک مانند خون، ادرار، خلط، شیر مادر، مایع مغزی- نخاعی و... شناسایی کرد. وزیکولهای خارج سلولی حاوی پروتئین، لیپید، DNA ، RNA ، محتوای ژنتیکی پاتوژنها مانند ویروسها و باکتریها و... میباشند (31). مولکولهای زیادی بر روی EVsها وجود دارد که میتوانند به تعداد زیادی از گیرندههای سلولی متصل شوند و یا از طریق فرآیند فاگوسیتوز سلولهای ایمنی محتویات وزیکولها وارد سلولهای هدف میشوند (32, 31). مطالعههای اخیر نشان دادهاند که سلولهای آلوده به ویروس یک سری وزیکولهای خارج سلولی تولید میکنند که حاوی پروتئینهای (gp61+++/Tax+++/HBZ+) و RNA ویروس میباشد. این وزیکولهای خارج سلولی قادر هستند بر روی سلولهای سالم تاثیر گذاشته و آنها را نیز درگیر کنند. همچنین در این مطالعه نشان داده شده، که میزان سنتز RNA ویروس HTLV-1 در موشهای NOG (موشهایی که زنجیره گاما رسپتور اینترلوکین 2 در آنها حذف شده است) مجاور شده با وزیکولهای خارج سلولی ویروس HTLV-1 در مقایسه با موشهای مجاور شده با وزیکولهای خارج سلولی سلولهای غیر عفونی افزایش یافته است.
همچنین میزان DNA ویروس در خون، ریه، طحال، کبد و مغز موشها پس از مجاور کردن با وزیکولهای HTLV-1 اندازهگیری گردید که افزایش سطح DNA ویروس در تمام بافتها به ویژه مغز مشاهده شد (33). مطالعهها نشان داده که وزیکولهای خارج سلولی از خون محیطی و مایع مغزی نخاعی بیماران مبتلا به HAM/TSP ، حاوی پروتئین ترانس اکتیو Tax میباشند که باعث افزایش تجمع سلولی و گسترش ویروس میشود (31). وزیکولهای خارج سلولی میتوانند باعث افزایش RNA و DNA پروویروس در خون، غدد لنفاوی و طحال شوند (34).
از آن جایی که مطالعههای زیادی بر روی وزیکولهای خارج سلولی مشتق از سلولهای آلوده به HTLV-1 صورت نگرفته است و مشخص نشده است که این EVs ها عفونی هستند یا خیر. بنابراین ضروری است وزیکولهای خارج سلولی تولید شده توسط سلولهای حاوی ویروس از نظر حضور ژنوم ویروس HTLV-1 ، پرووایرال لود ویروس، بیان ژنهای ویروسی و عفونی بودن مورد ارزیابی قرار گیرند.
نتیجهگیری
جهت تشخیص مولکولی ویروس HTLV-1 ، میتوان از DNA های پروویروس HTLV-1آزاد شده از سلولها در پلاسمای ناقلین بدون علامت در مواردی که سلولهای تک هستهای خون محیطی در دسترس نمیباشد، استفاده نمود. همچنین دیده شد که میزان بار ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت است که نشان دهنده تعداد کپی پایین ویروس HTLV-1 در پلاسمای این افراد میباشد.
حمایت مالی
این پروژه توسط مرکز تحقیقات انتقال خون، مؤسسه عالی آموزشی و پژوهشی طب انتقال خون تأمین مالی شده است.
ملاحظات اخلاقی
این مطالعه توسط کمیته اخلاق تحقیقات زیست پزشکی مؤسسه عالی آموزش و پژوهشی طب انتقال خون مورد بررسی و تائید قرار گرفت (IR.TMI.REC.1400.018).
عدم تعارض منافع
هیچگونه تعارض منافعی در مطالعه حاضر وجود نداشته
است.
نقش نویسندگان
منیره مسعودی: نوشتن مقاله و انجام آزمایشها، روششناسی، تحلیل و بررسی دادهها.
دکتر زهره شریفی: طراحی مطالعه، ویرایش مقاله، بررسی و
تفسیر دادهها، نظارت بر انجام آزمایشها
دکتر مهتاب مقصودلو: طراحی مطالعه، ویرایش مقاله و مشاوره آماری
تشکر و قدردانی
این پژوهش حاصل پایاننامه کارشناسی ارشد مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ایران میباشد. بدین وسیله از مؤسسه جهت حمایت مالی تشکر میگردد.
متن کامل: (816 مشاهده)
تشخیص مولکولی پروویروس HTLV-1 از DNA آزاد شده از سلولها در پلاسما،
در موارد عدم دسترسی به DNA ژنومی سلولهای تک هستهای خون محیطی
منیره مسعودی1، زهره شریفی2، مهتاب مقصودلو3
چکیده
سابقه و هدف
ویروس لنفوتروپیک تیپ I انسانی، عمدتاً لنفوسیتها را آلوده میکند. برای تشخیص آین ویروس ، عمدتاً سلولهای هسته دار مورد ارزیابی قرار میگیرد. با توجه به طول عمر کوتاه سلول ها، جمعآوری نمونه یک چالش به حساب میآید. هدف از این مطالعه، بررسی حضور ویروس HTLV-1 در DNA ژنومی آزاد شده از سلول ها در پلاسما و تعیین بار ویروسی آن در اهداکنندگان خون وسترن بلات مثبت بود.
مواد و روشها
در این مطالعه توصیفی، 30 اهداکننده ناقل ویروس HTLV-1 وسترن بلات مثبت بررسی شدند. با استفاده از کیت استخراج، DNA ژنومی از پلاسما، و PBMC جداسازی شده با فایکول، استخراج گردید. حضور ویروس HTLV-1 با آزمایش مولکولی Nested PCR تایید و جهت تعیین بار ویروس از آزمایش کمی Real-Time Sybergreen PCR استفاده گردید.
یافتهها
میانگین سنی افراد برابر با 5/13 ± 9/42 سال بود. 3/93% از افراد مذکر و 6/6% مؤنث بودند. ناحیه ژنی LTR به روش Nested PCR در80% از نمونههای پلاسما و 100% نمونههای PBMC شناسایی گردید. میانه لود ویروس در پلاسما برابر با Copies/µL 90/1 و میانه لود ویروس در PBMC برابر با PBMC 106/ Copies 20 بود.
نتیجه گیری
جهت تشخیص مولکولی ویروس HTLV-1 ، میتوان پروویروس HTLV-1 را در DNA آزاد شده از سلولها در پلاسما، در مواردی که سلولهای تک هستهای خون محیطی در دسترس نمیباشد، مورد استفاده قرار داد. همچنین دیده شد که میزان بار ویروس در پلاسمای ناقلین بدون علامت نسبت به PBMC حدود 10 برابر کمتر میباشد.
کلمات کلیدی: ویروس لنفوتروپیک انسانی تیپ I ، اسید نوکلئیک آزاد شده از سلول، PCR کمی
تاریخ دریافت: 12/12/1402
تاریخ پذیرش: 24/02/1403
1- دانشجوی کارشناسی ارشد خونشناسی و بانک خون ـ مرکز تحقیقات انتقال خونـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خونـ تهرانـ ایران
1- مؤلف مسئول: PhD ویروسشناسی ـ استاد مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران ـ صندوق پستی: 1157-14665
3- متخصص پزشکی اجتماعی ـ استاد مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
در موارد عدم دسترسی به DNA ژنومی سلولهای تک هستهای خون محیطی
منیره مسعودی1، زهره شریفی2، مهتاب مقصودلو3
چکیده
سابقه و هدف
ویروس لنفوتروپیک تیپ I انسانی، عمدتاً لنفوسیتها را آلوده میکند. برای تشخیص آین ویروس ، عمدتاً سلولهای هسته دار مورد ارزیابی قرار میگیرد. با توجه به طول عمر کوتاه سلول ها، جمعآوری نمونه یک چالش به حساب میآید. هدف از این مطالعه، بررسی حضور ویروس HTLV-1 در DNA ژنومی آزاد شده از سلول ها در پلاسما و تعیین بار ویروسی آن در اهداکنندگان خون وسترن بلات مثبت بود.
مواد و روشها
در این مطالعه توصیفی، 30 اهداکننده ناقل ویروس HTLV-1 وسترن بلات مثبت بررسی شدند. با استفاده از کیت استخراج، DNA ژنومی از پلاسما، و PBMC جداسازی شده با فایکول، استخراج گردید. حضور ویروس HTLV-1 با آزمایش مولکولی Nested PCR تایید و جهت تعیین بار ویروس از آزمایش کمی Real-Time Sybergreen PCR استفاده گردید.
یافتهها
میانگین سنی افراد برابر با 5/13 ± 9/42 سال بود. 3/93% از افراد مذکر و 6/6% مؤنث بودند. ناحیه ژنی LTR به روش Nested PCR در80% از نمونههای پلاسما و 100% نمونههای PBMC شناسایی گردید. میانه لود ویروس در پلاسما برابر با Copies/µL 90/1 و میانه لود ویروس در PBMC برابر با PBMC 106/ Copies 20 بود.
نتیجه گیری
جهت تشخیص مولکولی ویروس HTLV-1 ، میتوان پروویروس HTLV-1 را در DNA آزاد شده از سلولها در پلاسما، در مواردی که سلولهای تک هستهای خون محیطی در دسترس نمیباشد، مورد استفاده قرار داد. همچنین دیده شد که میزان بار ویروس در پلاسمای ناقلین بدون علامت نسبت به PBMC حدود 10 برابر کمتر میباشد.
کلمات کلیدی: ویروس لنفوتروپیک انسانی تیپ I ، اسید نوکلئیک آزاد شده از سلول، PCR کمی
تاریخ دریافت: 12/12/1402
تاریخ پذیرش: 24/02/1403
1- دانشجوی کارشناسی ارشد خونشناسی و بانک خون ـ مرکز تحقیقات انتقال خونـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خونـ تهرانـ ایران
1- مؤلف مسئول: PhD ویروسشناسی ـ استاد مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران ـ صندوق پستی: 1157-14665
3- متخصص پزشکی اجتماعی ـ استاد مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
مقدمه
ویروس لنفوتروپیک تیپ یک انسانی (HTLV-1) ، رتروویروس اگزوژن میباشد (1). این ویروس عضو خانواده رتروویریده و جنس دلتا ویروس است (3، 2). رتروویروسها RNA ویروسهایی هستند که فرم cDNA آنها به داخل ژنوم سلول میزبان اینتگره میشود (4). تعداد افراد مبتلا به ویروس HTLV-1 در سراسر جهان حدود 20-1 میلیون نفر تخمین زده شده است که 10-5 میلیون نفر در مناطق اندمیک زندگی میکنند (7-5، 3، 1). شیوع این ویروس در مناطق اندمیک مانند ژاپن، جنوب صحرای آفریقا، منطقه کارائیب و جنوب آمریکا زیاد میباشد. همچنین این ویروس به میزان کمتر در رومانی، ملانژی-استرالیا و ایران حضور دارد (3). در حال حاضر در ایران غربالگری ویروس HTLV-1 در اهداکنندگان خون، عمدتاً در 7 استان خراسان رضوی، خراسان شمالی، خراسان جنوبی، گلستان، البرز، اردبیل و آذربایجان شرقی انجام میشود (8).
این ویروس از سه طریق مادر به فرزند، تماس جنسی و به واسطه پزشکی از فرد به فرد دیگر منتقل میشود، که خطر انتقال ویروس از راه تزریق خون را میتوان با فیلتر کردن خون و فرآیند کاهش لکوسیت قبل از تزریق خون کاهش داد (9، 5، 3). احتمال تغییرات سرمی (Seroconversion) ویروس HTLV-1 از طریق تزریق خون حدود 60%-40% گزارش شده است. بنابراین خطر انتقال ویروس از راه تزریق خون در مناطقی با شیوع بالای ویروس حائز اهمیت باشد (10).
ویروس HTLV-1 عمدتاً سلولهای لنفوسیت TCD4+، که در ایجاد پاسخهای ایمنی دخیل هستند، را درگیر میکند (11). گسترش این ویروس عمدتاً از طریق تکثیر سلولهای آلوده از راه تماس سلول به سلول میباشد (12). در شرایط آزمایشگاهی مشاهده شده است که ویروس HTLV-1 ویریونهای آزاد کمی تولید میکند و بیشتر به صورت اینتگره درون ژنوم سلول میزبان میباشد (13). در مطالعههای اخیر ذکر شده است که ژنوم ویروس HTLV-1 به واسطه حضور DNA آزاد شده از سلولها در مایعات بیولوژیک بدن (cfDNA)، قابل شناسایی است.
cfDNAشامل قطعات اسیدنوکلئیک تکرشتهای و دو رشتهای خارج سلولی میباشد که عمدتاً ناشی از نکروز و آپوپتوز سلولی است. اخیراً دیده شده است که انتشار cfDNA از طریق میکرووزیکولهایی مانند اگزوزوم نیز امکانپذیر میباشد. cfDNA در پلاسما و مایعات بیولوژیک مانند CSF قابل شناسایی هستند. اسیدهای نوکلئیک درون زاد به طور دائم در جریان خون حضور دارند و مقدار آن در شرایط پاتولوژیک تغییر میکند. مطالعهها نشان دادهاند که میتوان از cfDNA به عنوان یک نشانگر زیستی برای تشخیص، تعیین اثر درمان و پیشآگهی بیماریها استفاده کرد (17-14).
در پژوهشهای مولکولی ویروس HTLV-1 ، عمدتاً ژنوم سلولهای تک هستهای خون محیطی و بافیکوت مورد ارزیابی قرار میگیرد. از آن جایی که طول عمر سلولهای هدف چند روز میباشد و این امکان وجود دارد که در روند جمعآوری و انتقال نمونه، سلولهای هستهدار از بین بروند، جمعآوری نمونه در مطالعههای مولکولی HTLV-1 یک چالش به حساب میآید. بنابراین اگر امکان تشخیص ژنوم ویروس HTLV-1 در نمونههای سرم و پلاسما، که شرایط نگهداری طولانی مدت دارند، وجود داشته باشد، تا حدودی میتوان این چالش را برطرف نمود. هدف از این مطالعه، بررسی حضور ویروس HTLV-1 در DNA آزاد شده از سلول در پلاسما و تعیین بار ویروسی آن در مقایسه با سلولهای تک هستهای خون محیطی (PBMC) در اهداکننـدگان خـون وستـرن بـلات مثبت بود.
مواد و روشها
این پژوهش از نوع توصیفی بود و جامعه مورد مطالعه شامل 30 اهداکننده خون ایرانی بودند که به مراکز انتقال خون کشور، به منظور غربالگری ویروس HTLV-1 مراجعه داشتند. معیار ورود افراد به این مطالعه حضور آنتی بادی علیه ویروس HTLV-1/2 (نتیجه Reactive آزمایش غربالگری CLEIA) و نتیجه مثبت آزمایش تائیدی وسترن بـلات و عـدم حضـور ویروسهـای منتقلـه از راه خــون
مانندHIV ، HCV و HBV تعیین شد.
جمعآوری نمونه:
در این مطالعه از نمونههای خون کامل استفاده شد. پس از جمعآوری، نمونهها به مدت 10 دقیقه با دور RPM 3000 سانتریفوژ شدند. لایه پلاسما جدا گردیده و در فریزر 30- درجه سانتیگراد نگهداری شد. به منظور جداسازی سلولهای تکهستهای خون محیطی از فایکول (ایران، mBio) و سانتریفیوژ شیب غلظت استفاده شد. با کمک لام نئوبار تعداد سلولهای تک هستهای خون محیطی شمارش گردید. جهت استخراج DNA ژنومی تعداد یک میلیون PBMC در حجم µL 200 تهیه شد. به منظور استخراج DNA آزاد شده از سلول در پلاسما، از µL 200 پلاسما استفاده گردید. DNA پروویروس توسط کیت استخراج اسیدهای نوکلئیک ویروسی ساخت شرکت دینابایو ایران با شماره کالانما 10025K طبق دستورالعمل شرکت سازنده استخراج گردید. غلظت و خلوص DNA استخراج شده با کمک دستگاه نانودراپ (آمریکا، دنوویکس) مورد ارزیابی قرار گرفت. نمونه DNA استخراج شده از پلاسما و PBMC تا زمان آزمایش در فریزر 30- درجه سانتیگراد نگهداری شدند. مابقی جهت نگهداری طولانی مدت در فریزر 70- درجه سانتیگراد نگهداری شدند.
تائید حضور ویروس به روش Nested PCR :
به منظور تائید حضور ویروس در نمونههای مورد استفاده، از آزمایش Nested PCR استفاده گردید. واکنش Nested PCR به منظور تکثیر ناحیه ژنی LTR ، بر روی DNA استخراج شده از پلاسما (cfDNA) و DNA ژنومی انجام شد. توالی آغازگر پیشبرنده بیرونی ناحیه ژنی LTR ، 5’ ACC ATG AGC CCC AAA TAT CCC C 3’ و توالی آغازگر معکوس بیرونی ناحیه ژنی LTR ، 5’ TCG TAT CCC GGA CGA GCC CCC AA 3’ بود. توالی آغازگر پیشبرنده درونی ناحیه ژنی LTR ، 5’ AGA CTA AGG CTC TGA CG TCT CCC 3’ و توالی آغازگر معکـوس درونـی ناحیـه ژنـی LTR ، 5’ AAT TTC TCT CCT GAG AGT GCT ATA G 3’ بود (18).

به جهت انجام واکنش Nested PCR ، µL 10 از (آلمان، آمپلیکون) X2 Master Mix ، µL 5/0 از آغازگرهای درونی و بیرونی (µL 10)، µL 4 از آب مقطر استریل و (ng 100-50) µL 5 از DNA استخراج شده از پلاسما و سلولهای تکهستهای خون محیطی استفاده شد. حجم نهایی PCR ، µL 20 تعیین شد. برنامه دمایی و زمانی واکنش Nested PCR در جدول آورده شده است (جداول 1 و 2). پس از اتمام واکنش، محصول مرحله دوم Nested PCR بر روی ژل آگارز 5/1% و رنگ DNA Green viewer در مقابل نشانگر وزن مولکولی bp 100 الکتروفورز انجام شد. سپس با دستگاه ژل داک (ایران، ATP) در مقابل نور فرا بنفش آشکارسازی گردید.
اندازهگیری بار ویروس HTLV-1 به روشQuantitative Real-Time PCR :
به منظور اندازهگیری میزان بار ویروس HTLV-1 از آزمایشQuantitative Real-Time PCR به روش سایبرگرین و ژن هدف Tax استفاده گردید. توالی آغازگر پیشبرنده ژن Tax، 5’ CCC ACA ATC CAA CCA GCT CAG 3’ و توالی آغازگر معکوس 5’ GTG GTG AAG CTG CCA TCG GGT TTT 3’ بود (19).
به جهت طبیعی کردن نتایج و کنترل داخلی، آزمایش PCR از ژن ERV3 استفاده شد. در سلولهای دیپلوئید

انسانی دو کپی از ژن ERV3 حضور دارد. توالی آغازگر پیشبرنده ژن ERV3 ، 5’ CAT GGG AAG CAA GGG AAC TAA TG 3’ و توالی آغازگر معکوس 5’ CCC AGC GAC CAA TAC AGA ATT T 3’ میباشد (20).
جهت انجام واکنش qPCR به روش سایبرگرین، µL 5/7 از (آلمان، آمپلیکون) X2 Master Mix حاوی رنگ سایبرگرین، µL 5/0 آغازگرهای ذکر شده، µL 5/4 آب مقطر استریل و حجم (ng 100-50) µL 2 ، DNA استخراج شده (gDNA و cfDNA) استفاده گردید. حجم نهایی واکنش qPCR، µL 15 تعیین شد. برنامه دمایی و زمانی مورد استفاده در جدول آورده شده است (جداول 4-2). جهت تعیین تعداد کپی ژن Tax ویروس، از پلاسمید حاوی ژن Tax ویروس HTLV-1 تحت عنوان نمونه استاندارد استفاده و منحنی استاندارد رسم گردید. مطابق فرمولHTLV DNA average copy number ERV DNA average copy number
آنالیز آماری:
جهت انجام تحلیل آماری این پژوهش از نرم افزار SPSS ورژن 26 استفاده شد.
یافتهها
مطالعه حاضر بر روی 30 اهداکننده خون ناقل ویروس HTLV-1 بدون علامت با نتیجه وسترن بلات مثبت انجام شد. 3/93% از افراد (28 نفر) مذکر و 6/6% از افراد (2 نفر) مؤنث بودند. میانگین سنی افراد مورد مطالعه برابر با 5/13 ± 9/42 سال بود. 90% از افراد (27 نفر) متأهل و 10% از افراد (3 نفر) مجرد بودند.
یافتههای بالینی و بار ویروسی:
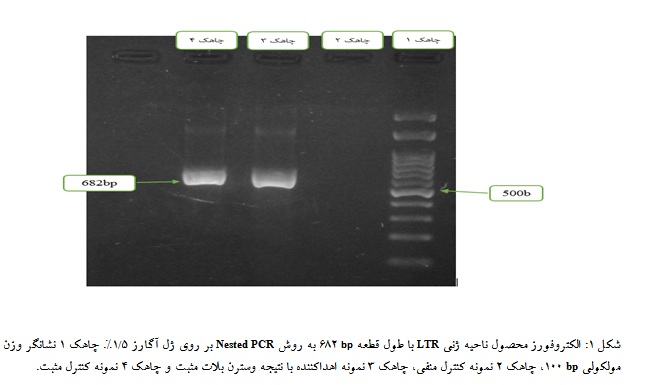
30 نمونه خون کامل با نتیجه وسترنبلات مثبت HTLV-1 جمعآوری گردید. با روش Nested PCR ناحیه ژنی LTR با طول قطعه bp 682 مورد ارزیابی قرار گرفت. این ناحیه ژنی تنها در 80% موارد (24 مورد) DNA آزاد شده از سلول در نمونه پلاسما شناسایی گردید. ناحیه ژنی LTR در 100% موارد (30 مورد) DNA ژنومی سلولهای تکهستهای خون محیطی به روش Nested PCR شناسایی گردید (شکل 1).
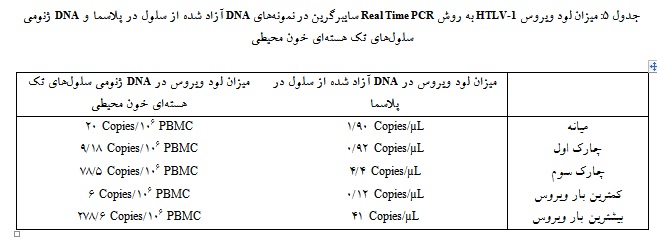
میزان بار ویروسی در این نمونهها با Real Time PCR به روش سایبرگرین با ژن هدف Tax اندازهگیری شد. میزان لود ویروس پلاسما (cfDNA) دارای توزیع غیر طبیعی بوده و میانه لود ویروس برابر با Copies/µL 90/1 بود (001/0 p<). چارک اول بار ویروس برابر با 92/0 Copies/µL و چارک سوم برابر با Copies/µL 4/4 بود (Copies/µL 48/3 IQR:). کمترین و بیشترین میزان لود ویروس به ترتیب برابر با Copies/µL12/0 و Copies/µL 41/0 تعیین شد. میزان لود ویروس سلولهای تکهستهای خون محیطی (gDNA) دارای توزیـع غیـر طبیعی بــوده و
جهت انجام واکنش qPCR به روش سایبرگرین، µL 5/7 از (آلمان، آمپلیکون) X2 Master Mix حاوی رنگ سایبرگرین، µL 5/0 آغازگرهای ذکر شده، µL 5/4 آب مقطر استریل و حجم (ng 100-50) µL 2 ، DNA استخراج شده (gDNA و cfDNA) استفاده گردید. حجم نهایی واکنش qPCR، µL 15 تعیین شد. برنامه دمایی و زمانی مورد استفاده در جدول آورده شده است (جداول 4-2). جهت تعیین تعداد کپی ژن Tax ویروس، از پلاسمید حاوی ژن Tax ویروس HTLV-1 تحت عنوان نمونه استاندارد استفاده و منحنی استاندارد رسم گردید. مطابق فرمول
آنالیز آماری:
جهت انجام تحلیل آماری این پژوهش از نرم افزار SPSS ورژن 26 استفاده شد.
یافتهها
مطالعه حاضر بر روی 30 اهداکننده خون ناقل ویروس HTLV-1 بدون علامت با نتیجه وسترن بلات مثبت انجام شد. 3/93% از افراد (28 نفر) مذکر و 6/6% از افراد (2 نفر) مؤنث بودند. میانگین سنی افراد مورد مطالعه برابر با 5/13 ± 9/42 سال بود. 90% از افراد (27 نفر) متأهل و 10% از افراد (3 نفر) مجرد بودند.
یافتههای بالینی و بار ویروسی:
30 نمونه خون کامل با نتیجه وسترنبلات مثبت HTLV-1 جمعآوری گردید. با روش Nested PCR ناحیه ژنی LTR با طول قطعه bp 682 مورد ارزیابی قرار گرفت. این ناحیه ژنی تنها در 80% موارد (24 مورد) DNA آزاد شده از سلول در نمونه پلاسما شناسایی گردید. ناحیه ژنی LTR در 100% موارد (30 مورد) DNA ژنومی سلولهای تکهستهای خون محیطی به روش Nested PCR شناسایی گردید (شکل 1).
میزان بار ویروسی در این نمونهها با Real Time PCR به روش سایبرگرین با ژن هدف Tax اندازهگیری شد. میزان لود ویروس پلاسما (cfDNA) دارای توزیع غیر طبیعی بوده و میانه لود ویروس برابر با Copies/µL 90/1 بود (001/0 p<). چارک اول بار ویروس برابر با 92/0 Copies/µL و چارک سوم برابر با Copies/µL 4/4 بود (Copies/µL 48/3 IQR:). کمترین و بیشترین میزان لود ویروس به ترتیب برابر با Copies/µL12/0 و Copies/µL 41/0 تعیین شد. میزان لود ویروس سلولهای تکهستهای خون محیطی (gDNA) دارای توزیـع غیـر طبیعی بــوده و


میانه لود ویروس برابر با PBMC 106/Copies 20 بود (001/0 p<). چارک اول بار ویروس برابر با PBMC 106/Copies 18/9 و چارک سوم برابر با PBMC 106/Copies 5/78 بود (PBMC 106/Copies 32/69 IQR:). کمترین و بیشترین میزان لود ویروس به ترتیب برابر با PBMC 106/Copies 6 و PBMC 106/Copies 78/6 تعیین شد (جدول 5).
بحث
در این مطالعه میزان پرووایرال لود HTLV-1 در پلاسما و سلولهای تک هستهای خون محیطی اهداکنندگان خون با نتیجه وسترن بلات مثبت به روش qPCR اندازهگیری شد تا آشکار شود در مواردی که سرم یا پلاسما به جای استفاده از سلولهای تک هستهای خون محیطی برای تشخیص ویروس به کار برده میشود، تا چه حد نتایج و لود ویروس با یکدیگر مشابهت دارد. در این مطالعه، میزان میانه بار ویروس در پلاسمای ناقلین بدون علامت Copies/µL90/1 بود و میانه لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت برابر با PBMC 106/Copies 20 بود که نشان میدهد میزان لود ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از بار ویروس در Genomic DNA است. نتایج حاصل از این پژوهش، با نتایج سایر مطالعههای انجام شده در دیگر کشورها در طی سالهای اخیر مطابقت دارد و بیانکننده این موضوع میباشد که ژنوم ویروس HTLV-1 در مایعات بیولوژیک بدن مانند پلاسما قابل شناسایی میباشد. Cell-free DNA ، قطعات DNA هستند که برای اولین بار در سال 1948 در کمپلکسهای ایمنی بیماران مبتلا به لوپوس اریتروماتوز در سیستم گردش خون توصیف شدند (22). اهمیت بالینی cfDNA زمانی مشخص شد که محققان تفاوتهایی بین ویژگیهای cfDNA افراد سالم و بیمار گزارش کردند (23). توماس جوریس و همکاران در کشور انگلستان، 6 فرد سالم به عنوان گروه کنترل، 25 ناقل بدون علامت ویروس HTLV-1 ، 21 بیمار میلوپاتی ناشی از ویروس HTLV-1 (HAM) و 25 بیمار مبتلا به لوسمی سلول T بالغین (ATL) را مورد ارزیابی قرار دادند. cfDNA از پلاسمای خون و Genomic DNA از سلولهای تکهستهای خون محیطی این افراد جدا شد. به منظور تائید حضور DNA انسانی از ژن بتاگلوبین و همچنین برای اندازهگیری بار ویروس از ژن Tax و qPCR به روش سایبرگرین استفاده کردند. پروویروس HTLV-1 در 85% از cfDNA ها شناسایی شد و نشان داده شد که عدم شناسایی پروویروس در cfDNA به دلیل لود پایین ویروس در gDNA سلولهای تکهستهای خون محیطی مرتبط است. در این مطالعه بیان گردید که DNA پروویروس یکی از اجزا cfDNA پلاسما در ناقلین HTLV-1 میباشد و میزان پرووایرال لود ویروس HTLV-1 در cfDNA پلاسما تقریبا 10 برابر کمتر از پرووایرال لود ویروس در gDNA سلولهای تکهستهای خون محیطی است (24). بیماری لوسمی سلول T بزرگسالان براساس ویژگیهای بالینی و شرح حال بیمار به چهار زیر گروه بالینی Acute ، Lymphoma ، Chronic و Smoldering تقسیم میگردد. هیروکاتسویا و همکاران در کشور ژاپن cfDNA را از پلاسما و gDNA را از PBMC ، 42 ناقل بدون علامت، 9 بیمار مبتلا به Smoldering ATL ، 7 بیمار مبتلا به Chronic ATL ، 5 بیمار مبتلا به Lymphoma ATL و 4 بیمار مبتلا به Acute ATL استخراج کردند و به روش Droplet digital PCR میزان پرووایرال لود ویروس اندازهگیری شد. میانگین لود ویروس در نمونههای cfDNA پلاسما در ناقلین بدون علامت Copies/mL 7/0 ، نوع copies/mL Smoldering 5/17، نوع copies/ml Chronic ATL 3/56، نوع copies/mL Lymphoma ATL 1650 و نوع copies/mL Acute ATL 625 گزارش شد. میانگین پرووایرال لود ویروس در نمونههای gDNA سلولهای تکهستهای خون محیطی در ناقلین بدون علامت 6% ، نوع Smoldering ATL 3/16% ، نوع Chronic ATL 76% ، نوع Lymphoma ATL 5/8% و نوع Acute ATL 3/69% تعیین گردید. در این مطالعه بیان شد که بیماران مبتلا به لوسمی سلول T بالغین نوع Lymphoma بیشترین مقدار بار ویروس را در cfDNA پلاسما و کمترین میزان پرووایرال لود را در PBMC دارند (25). دانیل روزا و همکاران در کشور برزیل 48 اهداکننده خون که از نظر حضور ویروس HTLV-1 مثبت گزارش شده بودند را مورد ارزیابی قرار دادند. این افراد براساس میزان درگیری عصبی به دو گروه تقسیم شدند. گروه 1 متشکل از 23 بیمار بدون درگیری عصبی و گروه 2 متشکل از 25 بیمار با نشانههایی از درگیری عصبی بودند. در این مطالعه میزان پرووایرال لود ویروس در پلاسما و مایع مغزی نخاعی به روش Real-Time SYBR Green PCR اندازهگیری شد.
نتایج نشان دادند که میزان لود ویروس در پلاسمای افراد گروه 1 و 2 از نظر آماری تفاوت چشمگیری نداشتند. در حالی که به طور چشمگیری میزان لود ویروس در مایع مغزی ـ نخاعی گروه 2 (همراه با درگیری عصبی) نسبت به گروه 1 (بدون درگیری عصبی) بالاتر بود (03/0 p=). این نتیجه میتواند تائیدکننده این موضوع باشد که میزان لود ویروس در بیماران پاراپارزی اسپاستیک گرمسیری همراه میلوپاتی ناشی از ویروس HTLV-1 (HAM/TSP) حدود 10 تا 100 برابر بیشتر از ناقلین بدون درگیری عصبی میباشد (26). سیلوا فورتادو و همکاران در کشور برزیل میزان پرووایرال لود ویروس HTLV-1 را در 75 ناقل بدون علامت و 78 بیمار HAM مورد ارزیابی قرار دادند. در این پژوهش DNA ویروس از خون محیطی استخراج شد و با روش Real-Time SYBR Green PCR و ژن هدف Pol و آلبومین میزان لود ویروس اندازه گیری شد. در این مطالعه عنوان شده است که میزان بار ویروس در بیماران HAM (cell 104/Copies 336 cell median: 104 Copies/ 711) حدود 6 برابر بیشتر از ناقلین بدون علامت (cell 104 Copies/ 336 cell median: 104 Copies/ 117) است (27).
در این مطالعه، در 80% موارد DNA ژنومی استخراج شده از پلاسمای اهداکنندگان خون با نتیجه وسترن بلات مثبت، ناحیه ژنی LTR ویروس به روش Nested PCR شناسایی گردید. فابیو کابرال و همکاران میزان لود ویروس HTLV-1 را در DNA ژنومی آزاد شده از سلول، از پلاسمای 150 فرد مبتلا به ویروس (123 ناقل بدون علامت و 27 بیمار HAM/TSP) با هدف قرار دادن ژن Pol و کنترل داخلی آلبومین به روش TaqMan Real-Time PCR ارزیابی کردند. نتایج نشان دادند که تکثیر ژن Pol در 4% ناقل بدون علامت و 26% بیمار HAM/TSP وجود دارد (28). مطالعهها نشان دادند که خون کامل و فرآوردههای سلولی و بافتی میتوانند منجر به انتقال ویروس HTLV-1 شوند، ولی تاکنون علیرغم حضور پروویروس در cfDNA در پلاسما، انتقال ویروس HTLV-1 از فرآوردههای بدون سلول گزارش نشده است که به دلیل عدم حضور تعداد کافی ویریون HTLV-1 عفونتزا در پلاسمای بیماران میباشد. پژوهش انجام شده در این زمینه نشان داده که تعداد بسیار کمی از پروویروس های آزاد شده از سلولهای آلوده به HTLV-1 در شرایط آزمایشگاهی عفونی هستند. تخمین زده شده است که از هر 106-105 ذره تولیدی، تنها یک مورد عفونتزای باشد (30، 29).
همچنین در مطالعههایی، ژنوم ویروس در وزیکولهای خارج سلولی در پلاسمای افراد نیز شناسایی گردیده است. اخیراً ساختارهای کوچک متصل به غشا تحت عنوان وزیکولهای خارج سلولی (EVs) به دلیل حمل پروتئینها و RNA ویروس در چندین عفونت ویروسی مانند HIV-1، HTLV-1 و ویروس Zika مورد توجه قرار گرفته است. در شرایط فیزیولوژیک و پاتولوژیک تقریباً تمام سلولها EVs ترشح میکنند که میتوان آن را در مایعات بیولوژیک مانند خون، ادرار، خلط، شیر مادر، مایع مغزی- نخاعی و... شناسایی کرد. وزیکولهای خارج سلولی حاوی پروتئین، لیپید، DNA ، RNA ، محتوای ژنتیکی پاتوژنها مانند ویروسها و باکتریها و... میباشند (31). مولکولهای زیادی بر روی EVsها وجود دارد که میتوانند به تعداد زیادی از گیرندههای سلولی متصل شوند و یا از طریق فرآیند فاگوسیتوز سلولهای ایمنی محتویات وزیکولها، وارد سلولهای هدف میشوند (32، 31). مطالعههای اخیر نشان دادهاند که سلولهای آلوده به ویروس یک سری وزیکولهای خارج سلولی تولید میکنند که حاوی پروتئینها (gp61+++/Tax+++/HBZ+) و RNA ویروس میباشد. این وزیکولهای خارج سلولی قادر هستند بر روی سلولهای سالم تاثیر گذاشته و آنها را نیز درگیر کنند. همچنین در این مطالعه نشان داده شده، که میزان سنتز RNA ویروس HTLV-1 در موشهای NOG (موشهایی که زنجیره گاما رسپتور اینترلوکین 2 در آنها حذف شده است) مجاور شده با وزیکولهای خارج سلولی ویروسHTLV-1 در مقایسه با موشهای مجاور شده با وزیکولهای خارج سلولی سلولهای غیر عفونی افزایش یافته است. همچنین میزان DNA ویروس در خون، ریه، طحال، کبد و مغز موشها پس از مجاور کردن با وزیکولهای HTLV-1 اندازهگیری گردید که افزایش سطح DNA ویروس در تمام بافتها به ویژه مغز مشاهده گردید (33). مطالعهها نشان داده که وزیکولهای خارج سلولی از خون محیطی و مایع مغزی نخاعی بیماران مبتلا به HAM/TSP ، حاوی پروتئین ترانس اکتیو Tax میباشند که باعث افزایش تجمع سلولی و گسترش ویروس میشود (31). وزیکولهای خارج سلولی میتوانند باعث افزایش RNA و DNA پروویروس در خون، غدد لنفاوی و طحال شوند (34). از آن جایی که مطالعههای زیادی بر روی وزیکولهای خارج سلولی مشتق از سلولهای آلوده به HTLV-1 صورت نگرفته است و مشخص نشده است که این EVs ها عفونی هستند یا خیر، بنابراین ضروری است وزیکولهای خارج سلولی تولید شده توسط سلولهای حاوی ویروس از نظر حضور ژنوم ویروس HTLV-1، پرووایرال لود ویروس، بیان ژنهای ویروسی و عفونی بودن مورد ارزیابی قرار گیرند.
در این مطالعه میزان پرووایرال لود HTLV-1 در پلاسما و سلولهای تکهستهای خون محیطی اهداکنندگان خون با نتیجه وسترن بلات مثبت به روش qPCR سایبرگرین اندازهگیری شد تا در مواردی که سرم یا پلاسما به جای استفاده از سلولهای تک هستهای خون محیطی برای تشخیص ویروس به کار برده میشود یا لود ویروس تعیین میشود، تا چه حد نتایج با یکدیگر مشابهت دارد. در این مطالعه، میزان میانه بار ویروس در پلاسمای ناقلین بدون علامت Copies/µL90/1 بود و میانه لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت برابر با PBMC 106 Copies/ 20 بود که نشان میدهد میزان لود ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از بار ویروس در Genomic DNA است. نتایج حاصل از این پژوهش، با نتایج سایر مطالعههای انجام شده در دیگر کشورها در طی سالهای اخیر مطابقت دارد و بیانکننده این موضوع میباشد که ژنوم ویروس HTLV-1 در مایعات بیولوژیک بدن مانند پلاسما قابل شناسایی میباشد. Cell-free DNA قطعات DNA هستند که برای اولین بار در سال 1948 در کمپلکسهای ایمنی بیماران مبتلا به لوپوس اریتروماتوز در سیستم گردش خون توصیف شدند (22). اهمیت بالینی cfDNA زمانی مشخص شد که محققان تفاوتهایی بین ویژگیهای cfDNA افراد سالم و بیمار گزارش کردند (23). توماس جوریس و همکاران در کشور انگلستان، 6 فرد سالم به عنوان گروه کنترل، 25 ناقل بدون علامت ویروس HTLV-1 ، 21 بیمار مبتلا به HAM و 25 بیمار مبتلا به ATL را مورد ارزیابی قرار دادند. cfDNA از پلاسمای خون و Genomic DNA از سلولهای تکهستهای خون محیطی این افراد جدا شد. به منظور تائید حضور DNA انسانی از ژن بتاگلوبین و همچنین برای اندازهگیری بارویروس از ژن Tax و qPCR به روش سایبرگرین استفاده کردند. پروویروس HTLV-1 در 85% از cfDNA شناسایی شد و نشان داده شد که عدم شناسایی پروویروس در cfDNA به لود پایین ویروس در gDNA سلولهای تکهستهای خون محیطی مرتبط است. در این مطالعه بیان گردید که DNA پروویروس یکی از اجزای cfDNA پلاسما در ناقلین HTLV-1 میباشد و میزان پرووایرال لود ویروس HTLV-1 در cfDNA پلاسما تقریباً 10 برابر کمتر از پرووایرال لود ویروس در gDNA سلولهای تکهستهای خون محیطی میباشد (24). بیماری لوسمی سلول T بزرگسالان براساس ویژگیهای بالینی و شرح حال بیمار به چهار زیرگروه بالینی Acute ، Lymphoma، Chronic و Smoldering تقسیم میگردد. هیرو کاتسویا و همکاران در کشور ژاپن cfDNA را از پلاسما و gDNA از PBMC ، 42 ناقل بدون علامت، 9 بیمار مبتلا به Smoldering ATL، 7 بیمار مبتلا بهChronic ATL ، 5 بیمار مبتلا به Lymphoma ATL و 4 بیمار مبتلا به Acute ATL استخراج کردند و به روش Droplet digital PCR میزان پرووایرال لود ویروس اندازهگیری شد. میانگین لود ویروس در نمونههای cfDNA پلاسما در ناقلین بدون علامت copies/mL 7/0، نوع Smoldering ATL copies/mL 5/17، نوع Chronic ATL copies/mL 3/56، نوع Lymphoma ATL copies/mL 1650 و نوع Acute ATL copies/mL 625 گزارش شد. میانگین پرووایرال لود ویروس در نمونههای gDNA سلولهای تکهستهای خون محیطی در ناقلین بدون علامت 6% ، نوع Smoldering ATL 3/16%، نوع Chronic ATL 76%، نوع Lymphoma ATL 5/8% و نوع Acute ATL 3/69% تعیین گردید. در این مطالعه بیان شد که بیماران مبتلا به لوسمی سلول T بالغین نوع Lymphoma بیشترین مقدار بار ویروس را در cfDNA پلاسما و کمترین میزان پرووایرال لود را در PBMC دارند (25). دانیل رزا و همکاران در کشور برزیل 48 اهداکننده خون که از نظر حضور ویروس HTLV-1 مثبت گزارش شده بودند را مورد ارزیابی قرار دادند. این افراد براساس میزان درگیری عصبی به دو گروه تقسیم شدند. گروه 1 متشکل از 23 بیمار بدون درگیری عصبی و گروه 2 متشکل از 25 بیمار با نشانه هایی از درگیری عصبی بودند. در این مطالعه میزان پرووایرال لود ویروس در پلاسمـا و مایـع مغـزی نخاعـی بــه روش Real-Time
SYBR Green PCR اندازهگیری شد.
نتایج نشـان دادنـد کـه میـزان لود ویروس در پلاسمای
افراد گروه 1 و 2 از نظر آماری تفـاوت چشمگیـری نـداشتند. در حالی که به طور چشمگیری میزان لود ویروس در مایع مغزی نخاعی گروه 2 (همراه با درگیری عصبی) نسبت به گروه 1 (بدون درگیری عصبی) بالاتر بود (03/0 p=). این نتیجه میتواند تائیدکننده این موضوع باشد که میزان لود ویروس در بیماران HAM/TSP حدود 10 تا 100 برابر بیشتر از ناقلین بدون درگیری عصبی میباشد (26). سیلوا فورتادو و همکاران در کشور برزیل میزان پرووایرال لود ویروس HTLV-1 را در 75 ناقل بدون علامت و 78 بیمار HAM مورد ارزیابی قرار دادند. در این پژوهش DNA ویروس از خون محیطی استخراج شد و با روش Real-Time SYBR Green PCR و ژن هدف Pol و آلبومین میزان لود ویروس اندازهگیری شد. در این مطالعه عنوان شده است که میزان بار ویروس در بیماران HAM (cell 104Copies/ 336: cell median 104/Copies 711) حدود 6 برابر بیشتر از ناقلین بدون علامت (HAM 104 Copies/ 48 cell median: 104 Copies 117) است (27).
در این مطالعه، در 80% موارد DNA ژنومی استخراج شده از پلاسمای اهداکنندگان خون با نتیجه وسترن بلات مثبت ، ناحیه ژنی LTR ویروس به روش Nested PCR شناسایی گردید. فابیو کابرال و همکاران میزان لود ویروس HTLV-1 را در DNA ژنومی آزاد شده از سلول، از پلاسمای 150 فرد مبتلا به ویروس (123 ناقل بدون علامت و 27 بیمار HAM/TSP) با هدف قرار دادن ژن Pol و کنترل داخلی آلبومین به روش TaqMan Real-Time PCR ارزیابی کردند. نتایج نشان دادند که تکثیر ژن Pol در 4% ناقل بدون علامت و 26 بیمار HAM/TSP وجود دارد (28). مطالعهها نشان دادند که خون کامل و فرآوردههای سلولی و بافت میتوانند منجر به انتقال ویروس HTLV-1 شوند، ولی تاکنون علیرغم حضور پروویروس در cfDNA در پلاسما، انتقال ویروس HTLV-1 از فرآوردههای بدون سلول گزارش نشده است که به دلیل عدم حضور تعداد کافی ویریون HTLV-1 عفونت زا در پلاسمای بیماران میباشد. پژوهش انجام شده در این زمینه نشان داده که تعداد بسیار کمی از پروویروس های آزاد شده از سلولهای آلوده به HTLV-1 در شرایط آزمایشگاهی عفونی هستند. تخمین زده شده است که از هر 106-105 ذره تولیدی تنها یک مورد عفونتزا باشد (30، 29).
همچنین در مطالعههایی، ژنوم ویروس در وزیکولهای خارج سلولی در پلاسمای افراد نیز شناسایی گردیده است. اخیراً ساختارهای کوچک متصل به غشا تحت عنوان وزیکولهای خارج سلولی (EVs) به دلیل حمل پروتئینها و RNA ویروس در چندین عفونت ویروسی مانند HIV-1، HTLV-1 و ویروس Zika مورد توجه قرار گرفته است. در شرایط فیزیولوژیک و پاتولوژیک تقریبا تمام سلولها EVs ترشح میکنند که میتوان آن را در مایعات بیولوژیک مانند خون، ادرار، خلط، شیر مادر، مایع مغزی- نخاعی و... شناسایی کرد. وزیکولهای خارج سلولی حاوی پروتئین، لیپید، DNA ، RNA ، محتوای ژنتیکی پاتوژنها مانند ویروسها و باکتریها و... میباشند (31). مولکولهای زیادی بر روی EVsها وجود دارد که میتوانند به تعداد زیادی از گیرندههای سلولی متصل شوند و یا از طریق فرآیند فاگوسیتوز سلولهای ایمنی محتویات وزیکولها وارد سلولهای هدف میشوند (32, 31). مطالعههای اخیر نشان دادهاند که سلولهای آلوده به ویروس یک سری وزیکولهای خارج سلولی تولید میکنند که حاوی پروتئینهای (gp61+++/Tax+++/HBZ+) و RNA ویروس میباشد. این وزیکولهای خارج سلولی قادر هستند بر روی سلولهای سالم تاثیر گذاشته و آنها را نیز درگیر کنند. همچنین در این مطالعه نشان داده شده، که میزان سنتز RNA ویروس HTLV-1 در موشهای NOG (موشهایی که زنجیره گاما رسپتور اینترلوکین 2 در آنها حذف شده است) مجاور شده با وزیکولهای خارج سلولی ویروس HTLV-1 در مقایسه با موشهای مجاور شده با وزیکولهای خارج سلولی سلولهای غیر عفونی افزایش یافته است.
همچنین میزان DNA ویروس در خون، ریه، طحال، کبد و مغز موشها پس از مجاور کردن با وزیکولهای HTLV-1 اندازهگیری گردید که افزایش سطح DNA ویروس در تمام بافتها به ویژه مغز مشاهده شد (33). مطالعهها نشان داده که وزیکولهای خارج سلولی از خون محیطی و مایع مغزی نخاعی بیماران مبتلا به HAM/TSP ، حاوی پروتئین ترانس اکتیو Tax میباشند که باعث افزایش تجمع سلولی و گسترش ویروس میشود (31). وزیکولهای خارج سلولی میتوانند باعث افزایش RNA و DNA پروویروس در خون، غدد لنفاوی و طحال شوند (34).
از آن جایی که مطالعههای زیادی بر روی وزیکولهای خارج سلولی مشتق از سلولهای آلوده به HTLV-1 صورت نگرفته است و مشخص نشده است که این EVs ها عفونی هستند یا خیر. بنابراین ضروری است وزیکولهای خارج سلولی تولید شده توسط سلولهای حاوی ویروس از نظر حضور ژنوم ویروس HTLV-1 ، پرووایرال لود ویروس، بیان ژنهای ویروسی و عفونی بودن مورد ارزیابی قرار گیرند.
نتیجهگیری
جهت تشخیص مولکولی ویروس HTLV-1 ، میتوان از DNA های پروویروس HTLV-1آزاد شده از سلولها در پلاسمای ناقلین بدون علامت در مواردی که سلولهای تک هستهای خون محیطی در دسترس نمیباشد، استفاده نمود. همچنین دیده شد که میزان بار ویروس در پلاسمای ناقلین بدون علامت حدود 10 برابر کمتر از لود ویروس در سلولهای تکهستهای خون محیطی ناقلین بدون علامت است که نشان دهنده تعداد کپی پایین ویروس HTLV-1 در پلاسمای این افراد میباشد.
حمایت مالی
این پروژه توسط مرکز تحقیقات انتقال خون، مؤسسه عالی آموزشی و پژوهشی طب انتقال خون تأمین مالی شده است.
ملاحظات اخلاقی
این مطالعه توسط کمیته اخلاق تحقیقات زیست پزشکی مؤسسه عالی آموزش و پژوهشی طب انتقال خون مورد بررسی و تائید قرار گرفت (IR.TMI.REC.1400.018).
عدم تعارض منافع
هیچگونه تعارض منافعی در مطالعه حاضر وجود نداشته
است.
نقش نویسندگان
منیره مسعودی: نوشتن مقاله و انجام آزمایشها، روششناسی، تحلیل و بررسی دادهها.
دکتر زهره شریفی: طراحی مطالعه، ویرایش مقاله، بررسی و
تفسیر دادهها، نظارت بر انجام آزمایشها
دکتر مهتاب مقصودلو: طراحی مطالعه، ویرایش مقاله و مشاوره آماری
تشکر و قدردانی
این پژوهش حاصل پایاننامه کارشناسی ارشد مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ایران میباشد. بدین وسیله از مؤسسه جهت حمایت مالی تشکر میگردد.
نوع مطالعه: پژوهشي |
موضوع مقاله:
ويروس شناسي
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |






