جلد 18، شماره 4 - ( زمستان 1400 )
جلد 18 شماره 4 صفحات 278-269 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Taghikhani F, Mohammadipour M, Mousavi Hosseini K. Expression of recombinant Stem Cell Factor fused with Leucine Zipper. bloodj 2021; 18 (4) :269-278
URL: http://bloodjournal.ir/article-1-1333-fa.html
URL: http://bloodjournal.ir/article-1-1333-fa.html
تقی خانی فاطمه، محمدی پور مهشید، موسوی حسینی کامران. بیان فاکتور سلول بنیادی نوترکیب متصل به زیپلوسین. فصلنامه پژوهشی خون. 1400; 18 (4) :269-278
مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون
متن کامل [PDF 615 kb]
(1045 دریافت)
| چکیده (HTML) (1883 مشاهده)
مقدمه
فاکتور سلول بنیادیSCF:Stem Cell Factor)) با نامهای دیگری چون(SLF : Steel Factor)، (MGF : Mast Cell Growth Factor) و (KL : Kit ligand) نیز شناخته میشود(1). SCF فاکتور رشدی است که در مراحل اولیه نمو(HSCs : Hematopoietic stem cells) نقش دارد. SCF علاوه بر HSCs باعث تحریک، مهاجرت، بقا و تمایز پیشسازهای ملانوسیتها، ماستسلها و سلولهای زایا شده و سیگنالینگ گیرنده آن (c-Kit) در تشکیل طبیعی خون نقش دارد. این سیتوکاین گلیکوپروتئینی، توسط مغز استخوان تولید شده و توسط انواع مختلف سلولهای فیبروبلاست(شامل سلولهای استرومال مغز استخوان) و سلولهای اندوتلیال در سرتاسر بدن بیان میگردد. گلیکوزیلاسیون نقشی در فعالیت زیستی SCFندارد(3، 2).
SCFبه دو فرم فعال محلول و متصل به غشا وجود دارد و از نظر کمی و کیفی در نحوه سیگنالدهی متفاوت است(5-2). در حالی که فرم متصل به غشا SCF نقش بسیار مهمی در بدن ایفا میکند، فرم محلول آن جهت کشت و توسعه سلولهای بنیادی خونساز و جنینی استفاده میشود. انواع تجاری SCF همگی به فرم محلول تولید میشوند. انتظار میرود هر آن چه باعث افزایش عملکرد و پایداری SCF گردد، در افزایش تحریک تکثیر، مهاجرت، بقا و تمایز پیشسازهای سلولهای بنیادی خونساز، ملانوسیتها، ماستسلها و سلولهای زایا نیز اثری مطلوب بر جای بگذارد. این مهم در مطالعهای با ساخت SCF نوترکیب به شکل همودایمر کووالان فراهم گشته است(6). تولید SCF به صورت فیزیولوژیک به شکل مونومر بوده و به شکل همودایمر غیرکووالان واجد عملکرد میباشد و دایمریزاسیون آن نقش تنظیمی در تمایل اتصال و فعالیت گیرنده آن ایفا میکند(1). بر اساس مطالعه انجام شده فرم نوترکیب SCF ، ساختاری بسیار پایدارتر داشته و از نظر عملکرد قویتر از فرم طبیعی آن در تحریک ساخت سلولهای مورد مطالعه عمل میکند(6). از سویی دیگر با توجه به مطالعههای بسیاری که در آنها زیپلوسین به صورت نوترکیب یا طبیعی باعث دایمریزاسیون انواع مختلفی از پروتئینها شده، اثری مطلوب در عملکرد یا پایـداری آنهـا داشتـه اسـت(9-7). زیـپلوسیـن انتخابی
مناسب جهت دایمریزاسیون SCF به نظر میرسد. دلایل این ادعا در ساختار زیپلوسین و SCF نهفته است. زیپلوسین متشکل از دو هلیکس آلفا میباشد و مونومرهای آن با پیچش به دور هم با پیوندهای آبگریز مابین خود، تشکیل دایمر داده است و با تشکیل همودایمر، باعث همو و یا هترو دایمریزاسیون انواع بسیاری از پروتئینهای متصل به خودشان خواهد شد(11، 10). دو پروتومر تشکیلدهنده SCF نیز تحت زاویه 30 درجه، با اتصال سر به سر دایمر گشته و فقط به صورت دایمر دارای عملکرد میباشد(1). با بررسی روی مطالعههایی که در آنها زیپلوسین جهت دایمریزاسیون مونومرها به صورت نوترکیب یا طبیعی صورت گرفته است، به این نتیجه میتوان دست یافت که زیپلوسین از هر دو انتهای N و C ترمینال توالی خود، قابلیت اتصال به توالیهای دیگر جهت القای دایمریزاسیون را دارد(12-7). با توجه به ساختار SCF و زیپلوسین، از نظر فضایی امکان دایمریزاسیون SCF توسط زیپلوسین به صورت صحیح ممکن میباشد. جهت بهرهمندی از تمام مزایای میزبان بیانی پروکاریوتی از جمله امکان کشت و تولید در مقیاس وسیع با صرف هزینه و زمان کمتر در مقایسه با میزبانهای یوکاریوتی، از میزبان پروکاریوتی برای بیان SCF نوترکیب استفاده شد که در این راستا استفاده از باکتری اشریشیاکلای در مطالعهها در اولویت میباشد. SCFدارای دو پیوند دیسولفیدی در هر پروتومر خود میباشد که پیوند دیسولفیدی مابین سیستئین 4و89 جهت اتصال و فعالیت زیستی گیرنده ضروری بوده اما پیوند دیسولفیدی مابین سیستئین 43و138 برای اتصال گیرنده لازم نیست اما در فعالیت زیستی گیرنده نقش دارد؛ بر این اساس حضور هر دو پیوند برای دستیابی به عملکرد صحیح گیرنده ضروری است(3). اما از آن جا که محیط سیتوپلاسم باکتری(میزبان بیانی)، احیاکننده میباشد و امکان تشکیل باندهای دیسولفیدی در آن فراهم نیست، در این مطالعه از سویه مهندسی شده باکتری اشریشیاکلای بنام اریگامی(Origami) استفاده شد که انتظار میرود با دارا بودن جهشهایی در ژنهای تیوردکسینردوکتاز و گلوتاتیونردوکتاز، امکان تشکیل باندهای دیسولفیدی ضروری در ساختار پروتئین را فراهم کند و بهینهسازی توالی طراحی شده نیز برای این باکتری انجام گرفت.
مواد و روشها
مطالعه تجربی حاضر در مرکز تحقیقات سازمان انتقال خون تهران در سالهای 98-97 انجام شد که شامل دو بخش طراحی پروتئین نوترکیب توسط نرمافزارهای بیوانفورماتیک و تولید آزمایشگاهی پروتئین بود:
طراحی و پیشبینی ساختار اول، دوم و سوم پروتئین نوترکیب:
توالیهای SCF و زیپلوسین جهت طراحی پروتئین نوترکیب پس از بررسی پایگاههای اطلاعاتیNCBI, Uniprotkb و RCSB PDB و انجام بررسیها توسط برنامههای بیوانفورماتیکی انتخاب شدند(15-13). بررسی ساختار سه بعدی پروتئین نوترکیب طراحی شده LZ/SCF توسط برنامه Swissmodelانجام شد(16).
پیشبینی ساختار دوم، توسط نرمافزارPSIpred انجام شد(19-17). به منظور پیشبینی ساختار سوم و به دستآوردن شکل سه بعدی فیوژن پروتئین نوترکیب با فرمت فایل PDB، از نرمافزار Chimera استفاده شد. بر این اساس کاملترین ساختار متعلق به سازه طراحیشده متشکل از SCF متصل به زیپلوسین و فاقد لینکر میباشد(20).
بررسی پایداری ساختار:
بررسی پایداری ساختار پیشبینی شده پروتئین نوترکیب توسط نرمافزار RAMPAGE صورت گرفت(21).
انتخاب میزبان کلونینگ و بیان و بهینهسازی:
با توجه به ویژگیهای پروتئین نوترکیب، از باکتری TOP10 به عنوان میزبان کلونینگ و از باکتری اریگامی به عنوان میزبان بیانی استفاده شد و بهینهسازی توالی نوکلئوتیدی جهت بیان در میزبان پروکاریوتی توسط نرمافزار Optimizer برای باکتری اریگامی انجام گرفت(23، 22). بر اساس ویژگیهای پروتئین نوترکیب و باکتریهای انتخاب شده جهت بیان و کلونینگ، وکتور pET32a انتخاب گردید. بررسی و ایجاد جایگاههای برش آنزیم محدودکننده توسط نرمافزار NEBcutter و در نهایت ساخت توالی ژن طراحیشده توسط GenScript (آلمان) درون وکتور pET32a انجام شد(24).
آمادهسازی و تراریختی میزبان کلونینگ:
سلولهایTOP10 جهت پذیرش وکتور با روش شیمیایی با استفاده از کلریدکلسیم مستعدسازی گشته و تراریختی و انتقال وکتورpET32a-LZ/SCF به درون سلولهای مستعد به منظور افزایش تعداد وکتور با استفاده از روش شوک حرارتی انجام شد.
استخراج وکتور نوترکیب و بررسی صحت همسانهسازی ژن هدف در وکتور:
پس از ایجاد کلنیهای اختصاصی، وکتور نوترکیب pET32a-LZ/SCF از باکتری TOP10 استخراج شد. بررسی الگوی هضم آنزیمی با استفاده از آنزیمهای محدودکنندهNcoI و XhoIجهت بررسی خلوص وکتور و صحت همسانهسازی ژن هدف در وکتور pET32a انجام شد. الکتروفورز ژل آگارز جهت بررسی نتیجه انجام گرفت.
بیان پروتئین نوترکیب LZ/SCF :
پس از آمادهسازی سلولهای باکتری اریگامی جهت پذیرش وکتور و تراریختی با وکتورpET32a-LZ/SCF ، پروتئین نوترکیب LZ/SCF در باکتری اریگامی بیان گردید. بررسی بیان پروتئین نوترکیب توسط روش SDS–PAGE صورت گرفت. برای تأیید هویت پروتئین نوترکیب، وسترنبلات انجام گرفت.
یافتهها
طراحی و پیشبینی ساختار اول، دوم و سوم پروتئین نوترکیب:
در گام نخست توالی آمینواسیدی فاکتـور سلـول بنیادی
(SCF) و توالـی زیـپلوسیـن از بانـک اطلاعـات پروتئین
Uniprotkb به دست آمد. از توالی SCF(165) برای طراحی پروتئین نوترکیب استفاده شد که پیوندهای دیسولفیدی لازم برای عملکرد صحیح پروتئین و نقاط اتصال به گیرنده SCF (c-Kit) نیز در این محدوده توالی وجود دارد. پس از بررسی ساختار توالیهای زیپلوسین در اتصال با پروتئین SCF، توالی زیپلوسین مربوط به پروتئین Myocilin انتخاب گردید.
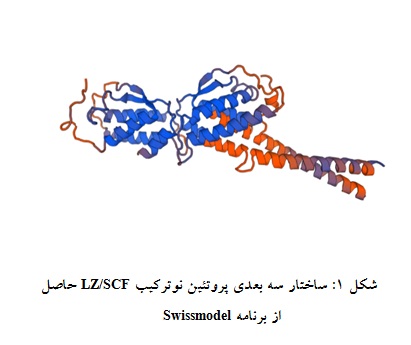
در طراحی ساختمان اول پروتئین نوترکیب، زیپلوسین برگرفته از پروتئین میوسیلین، Nترمینال و توالی مربوط به SCF، Cترمینال پروتئین طراحیشده را تشکیل میدهد. ساختار دوم پروتئین نوترکیب توسط نرمافزار PSIpred بررسی گشته و نتایج مدلسازی همولوژیکی حاصل از برنامه SWISS-MODEL نشاندهنده ساختاری از پروتئین نوترکیب LZ/SCF میباشد که در آن نحوه اتصال توالی پروتئین Leucine Zipper و SCF و ساختمان سهبعدی پروتئیـن نوترکیـب بـه صـورت مطلوب میباشد(شکل 1).
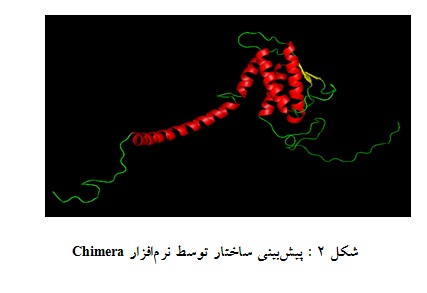
نتایج به دست آمده از پیشبینی ساختار سوم پروتئین نوترکیب LZ/SCF توسط نرمافزار Chimera به وسیله نرمافزارPyMOL مشاهده شد. مطابق انتظار در پروتئین نوترکیب، دومین تشکیلدهنده SCF واجد چهار هلیکس و دو beta-sheet و زیپلوسین نیز متشکل از یک هلیکس خطی میباشد(شکل 2).
بررسی پایداری ساختار:
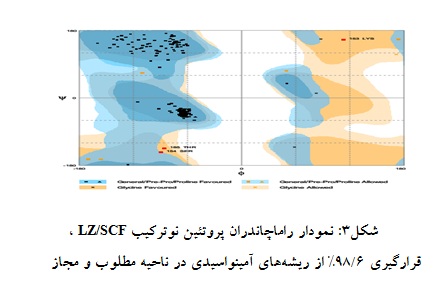
پایداری ساختار سوم پیشبینی شده برای پروتئین نوترکیب با استفاده از نمودار راماچاندران، نشاندهنده قرارگیری 8/95% از ریشههای آمینواسیدی در ناحیه مطلوب و 8/2% در ناحیه مجاز است در نتیجه پروتئین نوترکیب LZ/SCF از پایداری مناسبی در محیط سلول برخوردار میباشد(شکل 3).
بهینهسازی توالی نوکلئوتیدی:
به منظور بهینهسازی توالی پروتئین نوترکیب طراحی شده جهت بیان در میزبان پروکاریوتی(E. coli K12) از نرمافزار آنلاین Optimizer استفاده شد.
با توجه به نتایج حاصل از نرمافزار NEBcutter، آنزیمهای محدودکننده NcoI و XhoIانتخاب شدند. توالی محل اثر برش این آنزیمها به سکانس نوکلئوتیدی طراحی شده اضافه گشت. در نهایت توالی ژنی ساخته شده (GenScript، آلمان) و به درون وکتور pET32a وارد شد.
آمادهسازی و تراریختی میزبان کلونینگ:
پس از آمادهسازی سلولهای باکتری TOP10 (انستیتو پاستور، ایران) جهت پذیرش وکتور و نیز تراریختی این باکتریها با وکتور نوترکیب، باکتریها در محیط کشت انتخابی LB agar (Luria Bertani Broth) جامد حاوی آنتیبیوتیک آمپیسیلین (ژن مقاومت به آمپیسیلین بر روی وکتور pET32a-LZ/SCF وجود دارد) کشت و به صورت کلنی ظاهر شدند. این امر نشاندهنده موفقیت در تراریختی باکتریهای TOP10 با وکتور نوترکیب pET32a-LZ/SCF و ورود آن به ژنوم باکتری میباشد.
استخراج وکتور نوترکیب و بررسی صحت همسانهسازی ژن هدف در وکتور:
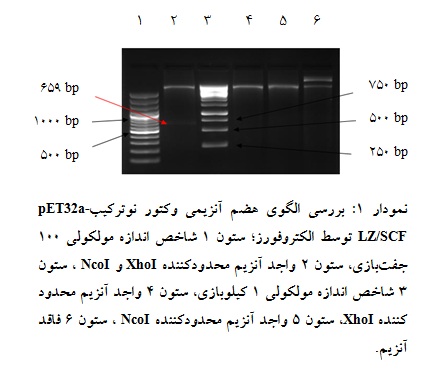
استخراج وکتور نوترکیب pET32a-LZ/SCF از باکتریهای E.coli سویه TOP10 با استفاده از کیت استخراج وکتور(Favorgene ، تایوان) به روش Miniprep انجام شد. جهت بررسی الگوی هضم آنزیمی، وکتور نوترکیب با استفاده از آنزیمهای محدودکننده NcoI و XhoI در بافر مشترک(Tango 2X) شکسته شد. بررسی کیفیت وکتور استخراج شده با استفاده از الکتروفورز ژل آگارز 1 درصد انجام شد. بررسی اندازه قطعات حاصل از هضم آنزیمی و حضور قطعه ژنی با اندازه مورد نظر، صحت همسانهسازی را تأیید نمود(نمودار 1).
آمادهسازی و تراریختی میزبان
 بیانی:
بیانی:
مستعدسازی سلولهای باکتری اریگامی(انستیتو پاستور، ایران) جهت پذیرش وکتور با استفاده از کلریدکلسیم و تراریختی و انتقال وکتورpET32a-LZ/SCF به داخل سلولهای مستعد شده توسط شوک حرارتی انجام شد. سپس باکتریها در محیط کشت انتخابی LB agar جامد حاوی آنتیبیوتیک آمپیسیلین و آنتیبیوتیکهای کانامایسین و تتراسایکلین (ژنهای مقاوم به این آنتیبیوتیکها در باکتری اریگامی وجود دارد) رشد کرده و کلنی تشکیل دادند که نشاندهنده موفقیت در تراریختی باکتریهای اریگامی با وکتور نوترکیب pET32a- LZ/SCF
و ورود آن به ژنوم باکتری است.
بیان پروتئین نوترکیب LZ/SCF :
جهت بیان پروتئین نوترکیب LZ/SCFپس از گرماگذاری کشتها به مدت یک شب در انکوباتور، از آن کشت تازه تهیه شد و القای بیان پروتئین نوترکیب LZ/SCF توسط IPTG (سیگما، آمریکا) انجام شد. بررسی بیان فیوژن پروتئین نوترکیب LZ/SCFدر اریگامی، توسط الکتروفورز پروتئینها با استفاده از روش SDS-PAGE انجام شد. نتایج حاصل از الکتروفورز(قسمت الف نمودار 2) نشاندهنده بیان پروتئین نوترکیب LZ/SCFدر ستون 2 پس از گذشت 6 ساعت پس از القا توسط IPTG در مقایسه با نمونههای قبل از القا در ستون 3 میباشد. در این میان در بررسی فواصل زمانی 2، 4 و 6 ساعت پس از القا با یکدیگر به نظر میرسد که باکتری در زمان 6 ساعت پس از القا توسط IPTG ، بیشترین میزان بیان پروتئین نوترکیب LZ/SCF را نشان داده و زمان مناسبتری برای برداشت پروتئین است(قسمت ب نمودار 2).
از میان کلنیهای تراریخت شده باکتری اریگامی با وکتور نوترکیب PET32a-LZ/SCF ، چهار کلنی مجزا و کوچک انتخاب شده و جهت بررسی بهترین غلظــت IPTG جهـت القـای بیان باکتری، از غلظتهای متفاوت IPTG 1/0- 5/0- 1- 2 میلیمولار استفاده شد که غلظت 1 میلیمولار بهترین میزان غلظت برای القای باکتری جهت بیان پروتئین نوترکیب LZ/SCF میباشد(قسمت ج نمودار 2).

 بهترین دانسیته جذب نوری ((OD رشد باکتری در طول موج ۶۰۰ نانومتر، حدود 5/0 ارزیابی گردید(قسمت د نمودار 2).
بهترین دانسیته جذب نوری ((OD رشد باکتری در طول موج ۶۰۰ نانومتر، حدود 5/0 ارزیابی گردید(قسمت د نمودار 2).
تأیید هویت پروتئین نوترکیب LZ/SCF :
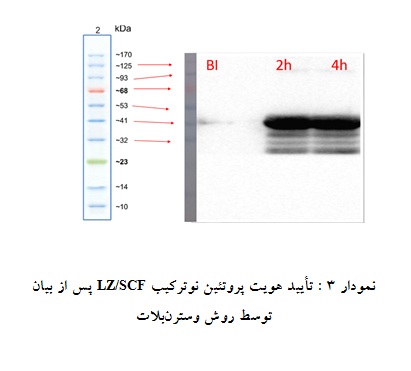
در انتها به دلیل این که توالی پروتئین نوترکیب LZ/SCF دارای His-Tag میباشد، پس از بیان هویت آن توسط وسترنبلات با استفاده از آنتیبادیهای مونوکلونال Anti His-Tag متصل به آنزیم HRP تأیید شد(نمودار 3).
بحث
نظـر به اهمیت حضور SCF در بقا، تکثیر و تمایز انواع
مختلـف سلـــولهـای بنیـادی خونسـاز، پیـشســازهای
ملانوسیتها، ماستسلها و سلولهای زایا و در راستای پیشرفت روزافزون بیوتکنولوژی و ژندرمانی در دهههای اخیر، SCF به عنوان یکی از گزینههای مهم در فعالیتهای بیوتکنولوژی شناخته شده است. SCF به نحو گستردهای در مطالعههای سلولهای بنیادی، بررسی روند مولکولی سرطانها و مهاجرت سلولهای سرطانی و مطالعههای مولکولی در انواع کمخونیها و حتی ایجاد روشهای درمانی جدید مورد استفاده است. بدین منظور در بسیاری از مطالعههای موجود در این حیطه، تولید SCF به شیوه نوترکیب مورد توجه قرار گرفته است. اما در این مسیر، صرف دستیابی به SCFنوترکیب اغناکننده و به صرفه نمیباشد؛ بلکه تولید SCF نوترکیبی که واجد ساختاری پایدارتر و عملکردی بیشتر از فرم طبیعی باشد، مطلوبتر خواهد بود.
در طی مطالعهای در سال 1997 مقایسه دو فرم دایمر کووالان (فرم نوترکیب) و غیرکووالان (فرم طبیعی) SCF نشاندهنده افزایش در تعداد کلنیهای مشاهده شده از SCF نوترکیب و توانایی تحریک رشد سلولهای مشتق از میزبانهای مورد نظر مطالعه و نیز پایداری بالاتر در مقایسه با SCF طبیعی بود(5). با توجه به این مطالعه که سعی بر ارتقای SCF از طریق تأثیر بر نحوه دایمریزاسیون مونومرهای SCF شده و باعث به دست آمدن راندمان بالاتری در بیان و پایداری SCF گشته است؛ ایده ارتقای SCF توسط تقویت و یا تغییر نحوه دایمریزاسیون SCF شکل گرفت. در مطالعه عنوان شده با تداخل در ساختار فضایی پروتئین به نتیجه موردنظر دست یافتند؛ در نتیجه با توجه به این که زیپلوسین یکی از مواردی است که به صورت طبیعی قادر به دایمریزاسیون توالیهای متصل به خود میباشد و با توجه به مطالعههای بسیاری که در این زمینه انجام شده است و نظر به تأثیرات زیپلوسین در پایداری و افزایش فعالیت دومین کاتالیتیک، توالیهای پروتئینهایی که موتیف زیپلوسین به صورت اتصال طبیعی یا نوترکیب به آنها متصل گشته است(9-7). در مطالعه پیشرو با اضافه کردن موتیف زیپلوسین به توالی SCF ، در واقع از زیپلوسین به عنوان ابزاری طبیعی جهت دستیابی به نتایج موردنظر بدون تداخل در ساختمان فضایی پروتئین استفاده شد. در مطالعه انجام شده در سال 2005، دایمریزاسیون مونومرهای پروتئین توسط زیپلوسین موجود در ساختار آن نشاندهنده تأثیر زیپلوسین بر پایداری این پروتئین در انواع شرایط سخت بافری میباشد(7). در پژوهش صورت گرفته در سال 2000 نیز جهت بررسی اتصال زیپلوسین به صورت نوترکیب به پروتئینها، در in vivo و in vitro ، افزایش فعالیت دومین کاتالیتیک متصل به زیپلوسین (فرم نوترکیب) نسبت به دومین کاتالیتیک (فرم طبیعی) مشاهده گردید(9).
تاکنون مطالعههای صورت گرفته برSCF در ایران، حول محور کلون و بیان توالی SCF بوده است. در مطالعهای در سال 2014 در ایران، توالی کدکننده فرم محلول SCF را کلون نمودند اما در مورد بیان آن گزارشی ارائه نگردید(25). همچنین در مطالعهای در سال 2017 در جهت بررسی بیان SCF در ایران، توالی کدکننده مونومر SCF کلون و در سویه Rosetta-gami بیان شد(4).
هدف مطالعه حاضر با توجه به مطالعههای عنوان شده، طراحی و بیان توالی پروتئینی نوترکیب LZ/SCF بود. جهت تحقق هدف پیشرو پس از بررسی پایگاههای اطلاعاتی مختلف، توالی آمینواسیدی فاکتور سلول بنیادی (SCF) و توالی زیپلوسین مربوط به پروتئین میوسیلین به دست آمد. جهت بررسی ساختار دوم پروتئین نوترکیب از نرمافزار PSIpred استفاده شد. ساختمان سه بعدی پروتئین نوترکیب LZ/SCF توسط برنامه Swiss-model بررسی و در نتایج مدلسازی همولوژیکی، نحوه اتصال توالی پروتئین Leucine Zipper و SCF به هم و ساختمان سه بعدی آن به صورت مطلوب میباشد. بررسی ساختار سوم توالیهای انتخابی نیز تحت چیدمانهای متفاوت توسط نرمافزار Chimera انجام گرفت. نتایج به دست آمده از پیشبینی ساختار سوم پروتئین نوترکیب LZ/SCF توسط نرمافزار Chimera نشاندهنده فولد مناسب و تشکیل ساختار کامل سازه طراحی شده میباشد. پایداری ساختار سوم پروتئین نوترکیب LZ/SCF با استفاده از نمودار راماچاندران بررسی شـد کـه نشاندهنـده پایـداری مناســب پروتئین نوترکیب
LZ/SCF در محیط سلول است.
از آن جا که در مطالعه صورت گرفته در سال 2017 جهت بیان کدونهای نادر، از سویه Rosetta-gami به عنوان میزبان بیانی استفاده شد(4). در مطالعه حاضر به دلیل استفاده از سویه اریگامی به عنوان میزبان بیانی و بهینهسازی توالی برای میزبان انتخاب شده توسط نرمافزار آنلاین اپتیمایزر، تمامی کدونها در اریگامی بیان خواهند شد. با توجه به نتایج حاصل از نرمافزار NEBcutter، آنزیمهای محدودکننده NcoI و XhoIانتخاب شدند. توالی محل اثر برش این آنزیمها که برای بیان پروتئین در میزبان پروکاریوتی ضروری است، به سکانس نوکلئوتیدی طراحی شده اضافه گشت. در نهایت توالی ژنی توسط(GenScript، آلمان) سنتز و درون وکتور pET32a سابکلون گردید.
جهت همسانهسازی سازه ژنی از باکتری TOP10 (انستیتو پاستور، ایران) به عنوان میزبان کلونینگ استفاده شد. استخراج وکتور pET32a- LZ/SCF از میزبان کلونینگ با استفاده از کیت استخراج پلاسمید(Favorgene ، تایوان) به روش Miniprep انجام شد. به منظور مشاهده و اطمینان از صحت و خلوص وکتور استخراج شده، محصول واکنش هضم آنزیمی، با استفاده از الکتروفورز در ژل آگارز مورد بررسی قرار گرفت.
در مطالعههای پیشین پس از بیان پروتئین نوترکیب در باکتری E.coli ، نیاز به Refolding پروتئین نوترکیب جهت تشکیل پیوندهای دیسولفیدی بود(26). از آن جا که دو پیوند دیسولفیدی SCF جهت اتصال و فعالیت زیستی گیرنده ضروری است و محیط باکتری E.coli به دلیل احیاکننده بودن سبب شکست پیوندهای دیسولفیدی موجود در پروتئین نوترکیب LZ/SCF میگردد، در نتیجه در مطالعه حاضر از باکتری اریگامی استفاده شد که موتانت برگرفته از سویه E.coli K12 میباشد زیرا سویه مهندسیشده اریگامی با دارا بودن جهشهایی در ژنهای تیوردکسینردوکتاز و گلوتاتیونردوکتاز، امکان تشکیل باندهای دیسولفیدی ضروری در ساختار پروتئین را فراهم میآورد. در مطالعه انجام گرفته در سال 2015، جهت بیان پیوندهای دیسولفیدی، توالی تیوردوکسینردوکتاز را با وکتور، بیان همزمان(coexpression) نموده که در مطالعه
حاضر مرتفع نمودن این نیاز با اضافه نمودن توالی تیوردوکسینردوکتاز به توالی پروتئین نوترکیب طراحی شده انجام گرفت(27). لازم به ذکر است که SCF در سلولهای پستانداران به صورت N-glycosylate است با این وجود ثابت شده است که حضور یا عدم حضور کربوهیدرات در SCF برای فعالیت زیستی ضروری به نظر نمیرسد. به همین دلیل بیان پروتئین نوترکیب LZ/SCF در باکتری E. coli باعث اختلال در فعالیت زیستی آن نخواهد شد. در نهایت بیان فیوژن پروتئین LZ/SCF در میزبان بیانی اریگامی به روش SDS – PAGE بررسی شد و سپس با روش وسترنبلات هویت پروتئین تأیید گشت.
نتیجهگیری
نتایج حاصل از مطالعه حاضر، تأیید ساختار مناسب سازه طراحی شده توسط نرمافزارهای مورد استفاده و موفق بودن کلون وکتور نوترکیب pET32a-LZ/SCF در میزبان باکتریایی کلونینگ TOP10)) و حصول کلونهای اختصاصی میباشد. بیان پروتئین LZ/SCF در میزبان باکتریایی بیان اریگامی نیز با موفقیت انجام شد.
تشکر و قدردانی
این مقاله حاصل پایاننامه دانشجویی دوره کارشناسی ارشد رشته بیوتکنولوژی پزشکی مرکز تحقیقات مؤسسه عالی آموزشی و پژوهشی طب انتقال خون میباشد. بودجه این تحقیق توسط مؤسسه عالی آموزشی و پژوهشی طب انتقال خون تأمین گردیده و همه مراحل علمی پروژه در مرکز تحقیقات سازمان انتقال خون ایران انجام شده است. پروپوزال این پایاننامه با دریافت کد اخلاق IR.TMI.REC.1397.002 در کمیته تخصصی اخلاق در پژوهشهای زیست پزشکی تصویب شده است.
متن کامل: (1385 مشاهده)
بیان فاکتور سلول بنیادی نوترکیب متصل به زیپلوسین
فاطمه تقی خانی1، مهشید محمدیپور2، کامران موسوی حسینی3
چکیده
سابقه و هدف
فاکتور سلول بنیادی، یک سیتوکاین خونی است که از طریق تأثیر بر روی سلولهای خونساز در تمایز سلولهای پیشساز خونی، بقا، تکثیر و تمایز ماستسل و همچنین افزایش تکثیر و تهاجم سلولهای توموری نقش ایفا میکند. زیپ لوسین به صورت نوترکیب یا طبیعی با تشکیل همودایمر در ساختار خود، باعث همو و یا هترو دایمریزاسیون انواع بسیاری از پروتئینهای متصل به خود شده و میتواند اثری مطلوب در عملکرد یا پایداری آنها داشته باشد. مطالعه انجام شده سعی در دایمریزاسیون فاکتور سلولهای بنیادی نوترکیب و زیپلوسین و بیان آن دارد.
مواد و روشها
مطالعه حاضر از نوع تجربی بود. شامل بخش طراحی پروتئین نوترکیب توسط نرمافزارهای بیوانفورماتیک و بخش آزمایشگاهی میباشد. شاخصهای مورد توجه در بخش طراحی از جمله ساختارهای پروتئینی توسط نرمافزارهای مختلف شامل PSIpred وChimera و پایداری پروتئین نوترکیب توسط RAMPAGE بررسی شد. پس از تعیین جایگاههای برش آنزیمی توسط NEBcutter و بهینهسازی برای باکتری اریگامی توسط Optimizer ، ساخت توالی انجام شد. در ادامه کلون آن در باکتری TOP10 و بیان پروتئین در باکتری اریگامی انجام گرفت.
یافتهها
نتایج حاصل از مطالعههای بیوانفورماتیک روی پروتئین نوترکیب Leucine Zipper / SCF، در برگیرنده شاخصهای لازم جهت ساختار صحیح پروتئین و دارای امتیازی بالا از لحاظ پایداری در سلول زنده بود و در ادامه نتایج حاصل از بخش تجربی نشان از کلون و بیان موفق آن داشت.
نتیجه گیری
نتایج این مطالعه، همسانهسازی و بیان موفق پروتئین نوترکیب Leucine Zipper/SCF در میزبان اریگامی را تائید نمود.
کلمات کلیدی: Stem Cell Factor، Leucine Zipper، پروتئین نوترکیب، دایمریزاسیون
تاریخ دریافت: 28/11/1398
تاریخ پذیرش: 16/05/1400
1- کارشناس ارشد بیوتکنولوژی پزشکی ـ مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
فاطمه تقی خانی1، مهشید محمدیپور2، کامران موسوی حسینی3
چکیده
سابقه و هدف
فاکتور سلول بنیادی، یک سیتوکاین خونی است که از طریق تأثیر بر روی سلولهای خونساز در تمایز سلولهای پیشساز خونی، بقا، تکثیر و تمایز ماستسل و همچنین افزایش تکثیر و تهاجم سلولهای توموری نقش ایفا میکند. زیپ لوسین به صورت نوترکیب یا طبیعی با تشکیل همودایمر در ساختار خود، باعث همو و یا هترو دایمریزاسیون انواع بسیاری از پروتئینهای متصل به خود شده و میتواند اثری مطلوب در عملکرد یا پایداری آنها داشته باشد. مطالعه انجام شده سعی در دایمریزاسیون فاکتور سلولهای بنیادی نوترکیب و زیپلوسین و بیان آن دارد.
مواد و روشها
مطالعه حاضر از نوع تجربی بود. شامل بخش طراحی پروتئین نوترکیب توسط نرمافزارهای بیوانفورماتیک و بخش آزمایشگاهی میباشد. شاخصهای مورد توجه در بخش طراحی از جمله ساختارهای پروتئینی توسط نرمافزارهای مختلف شامل PSIpred وChimera و پایداری پروتئین نوترکیب توسط RAMPAGE بررسی شد. پس از تعیین جایگاههای برش آنزیمی توسط NEBcutter و بهینهسازی برای باکتری اریگامی توسط Optimizer ، ساخت توالی انجام شد. در ادامه کلون آن در باکتری TOP10 و بیان پروتئین در باکتری اریگامی انجام گرفت.
یافتهها
نتایج حاصل از مطالعههای بیوانفورماتیک روی پروتئین نوترکیب Leucine Zipper / SCF، در برگیرنده شاخصهای لازم جهت ساختار صحیح پروتئین و دارای امتیازی بالا از لحاظ پایداری در سلول زنده بود و در ادامه نتایج حاصل از بخش تجربی نشان از کلون و بیان موفق آن داشت.
نتیجه گیری
نتایج این مطالعه، همسانهسازی و بیان موفق پروتئین نوترکیب Leucine Zipper/SCF در میزبان اریگامی را تائید نمود.
کلمات کلیدی: Stem Cell Factor، Leucine Zipper، پروتئین نوترکیب، دایمریزاسیون
تاریخ دریافت: 28/11/1398
تاریخ پذیرش: 16/05/1400
1- کارشناس ارشد بیوتکنولوژی پزشکی ـ مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
2- مؤلف مسئول: PhD ژنتیک مولکولی ـ استادیار مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران ـ صندوق پستی: 1157-14665
3- دکترای تخصصی شیمی دارویی ـ دانشیار مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
3- دکترای تخصصی شیمی دارویی ـ دانشیار مرکز تحقیقات انتقال خون ـ مؤسسه عالی آموزشی و پژوهشی طب انتقال خون ـ تهران ـ ایران
مقدمه
فاکتور سلول بنیادیSCF:Stem Cell Factor)) با نامهای دیگری چون(SLF : Steel Factor)، (MGF : Mast Cell Growth Factor) و (KL : Kit ligand) نیز شناخته میشود(1). SCF فاکتور رشدی است که در مراحل اولیه نمو(HSCs : Hematopoietic stem cells) نقش دارد. SCF علاوه بر HSCs باعث تحریک، مهاجرت، بقا و تمایز پیشسازهای ملانوسیتها، ماستسلها و سلولهای زایا شده و سیگنالینگ گیرنده آن (c-Kit) در تشکیل طبیعی خون نقش دارد. این سیتوکاین گلیکوپروتئینی، توسط مغز استخوان تولید شده و توسط انواع مختلف سلولهای فیبروبلاست(شامل سلولهای استرومال مغز استخوان) و سلولهای اندوتلیال در سرتاسر بدن بیان میگردد. گلیکوزیلاسیون نقشی در فعالیت زیستی SCFندارد(3، 2).
SCFبه دو فرم فعال محلول و متصل به غشا وجود دارد و از نظر کمی و کیفی در نحوه سیگنالدهی متفاوت است(5-2). در حالی که فرم متصل به غشا SCF نقش بسیار مهمی در بدن ایفا میکند، فرم محلول آن جهت کشت و توسعه سلولهای بنیادی خونساز و جنینی استفاده میشود. انواع تجاری SCF همگی به فرم محلول تولید میشوند. انتظار میرود هر آن چه باعث افزایش عملکرد و پایداری SCF گردد، در افزایش تحریک تکثیر، مهاجرت، بقا و تمایز پیشسازهای سلولهای بنیادی خونساز، ملانوسیتها، ماستسلها و سلولهای زایا نیز اثری مطلوب بر جای بگذارد. این مهم در مطالعهای با ساخت SCF نوترکیب به شکل همودایمر کووالان فراهم گشته است(6). تولید SCF به صورت فیزیولوژیک به شکل مونومر بوده و به شکل همودایمر غیرکووالان واجد عملکرد میباشد و دایمریزاسیون آن نقش تنظیمی در تمایل اتصال و فعالیت گیرنده آن ایفا میکند(1). بر اساس مطالعه انجام شده فرم نوترکیب SCF ، ساختاری بسیار پایدارتر داشته و از نظر عملکرد قویتر از فرم طبیعی آن در تحریک ساخت سلولهای مورد مطالعه عمل میکند(6). از سویی دیگر با توجه به مطالعههای بسیاری که در آنها زیپلوسین به صورت نوترکیب یا طبیعی باعث دایمریزاسیون انواع مختلفی از پروتئینها شده، اثری مطلوب در عملکرد یا پایـداری آنهـا داشتـه اسـت(9-7). زیـپلوسیـن انتخابی
مناسب جهت دایمریزاسیون SCF به نظر میرسد. دلایل این ادعا در ساختار زیپلوسین و SCF نهفته است. زیپلوسین متشکل از دو هلیکس آلفا میباشد و مونومرهای آن با پیچش به دور هم با پیوندهای آبگریز مابین خود، تشکیل دایمر داده است و با تشکیل همودایمر، باعث همو و یا هترو دایمریزاسیون انواع بسیاری از پروتئینهای متصل به خودشان خواهد شد(11، 10). دو پروتومر تشکیلدهنده SCF نیز تحت زاویه 30 درجه، با اتصال سر به سر دایمر گشته و فقط به صورت دایمر دارای عملکرد میباشد(1). با بررسی روی مطالعههایی که در آنها زیپلوسین جهت دایمریزاسیون مونومرها به صورت نوترکیب یا طبیعی صورت گرفته است، به این نتیجه میتوان دست یافت که زیپلوسین از هر دو انتهای N و C ترمینال توالی خود، قابلیت اتصال به توالیهای دیگر جهت القای دایمریزاسیون را دارد(12-7). با توجه به ساختار SCF و زیپلوسین، از نظر فضایی امکان دایمریزاسیون SCF توسط زیپلوسین به صورت صحیح ممکن میباشد. جهت بهرهمندی از تمام مزایای میزبان بیانی پروکاریوتی از جمله امکان کشت و تولید در مقیاس وسیع با صرف هزینه و زمان کمتر در مقایسه با میزبانهای یوکاریوتی، از میزبان پروکاریوتی برای بیان SCF نوترکیب استفاده شد که در این راستا استفاده از باکتری اشریشیاکلای در مطالعهها در اولویت میباشد. SCFدارای دو پیوند دیسولفیدی در هر پروتومر خود میباشد که پیوند دیسولفیدی مابین سیستئین 4و89 جهت اتصال و فعالیت زیستی گیرنده ضروری بوده اما پیوند دیسولفیدی مابین سیستئین 43و138 برای اتصال گیرنده لازم نیست اما در فعالیت زیستی گیرنده نقش دارد؛ بر این اساس حضور هر دو پیوند برای دستیابی به عملکرد صحیح گیرنده ضروری است(3). اما از آن جا که محیط سیتوپلاسم باکتری(میزبان بیانی)، احیاکننده میباشد و امکان تشکیل باندهای دیسولفیدی در آن فراهم نیست، در این مطالعه از سویه مهندسی شده باکتری اشریشیاکلای بنام اریگامی(Origami) استفاده شد که انتظار میرود با دارا بودن جهشهایی در ژنهای تیوردکسینردوکتاز و گلوتاتیونردوکتاز، امکان تشکیل باندهای دیسولفیدی ضروری در ساختار پروتئین را فراهم کند و بهینهسازی توالی طراحی شده نیز برای این باکتری انجام گرفت.
مواد و روشها
مطالعه تجربی حاضر در مرکز تحقیقات سازمان انتقال خون تهران در سالهای 98-97 انجام شد که شامل دو بخش طراحی پروتئین نوترکیب توسط نرمافزارهای بیوانفورماتیک و تولید آزمایشگاهی پروتئین بود:
طراحی و پیشبینی ساختار اول، دوم و سوم پروتئین نوترکیب:
توالیهای SCF و زیپلوسین جهت طراحی پروتئین نوترکیب پس از بررسی پایگاههای اطلاعاتیNCBI, Uniprotkb و RCSB PDB و انجام بررسیها توسط برنامههای بیوانفورماتیکی انتخاب شدند(15-13). بررسی ساختار سه بعدی پروتئین نوترکیب طراحی شده LZ/SCF توسط برنامه Swissmodelانجام شد(16).
پیشبینی ساختار دوم، توسط نرمافزارPSIpred انجام شد(19-17). به منظور پیشبینی ساختار سوم و به دستآوردن شکل سه بعدی فیوژن پروتئین نوترکیب با فرمت فایل PDB، از نرمافزار Chimera استفاده شد. بر این اساس کاملترین ساختار متعلق به سازه طراحیشده متشکل از SCF متصل به زیپلوسین و فاقد لینکر میباشد(20).
بررسی پایداری ساختار:
بررسی پایداری ساختار پیشبینی شده پروتئین نوترکیب توسط نرمافزار RAMPAGE صورت گرفت(21).
انتخاب میزبان کلونینگ و بیان و بهینهسازی:
با توجه به ویژگیهای پروتئین نوترکیب، از باکتری TOP10 به عنوان میزبان کلونینگ و از باکتری اریگامی به عنوان میزبان بیانی استفاده شد و بهینهسازی توالی نوکلئوتیدی جهت بیان در میزبان پروکاریوتی توسط نرمافزار Optimizer برای باکتری اریگامی انجام گرفت(23، 22). بر اساس ویژگیهای پروتئین نوترکیب و باکتریهای انتخاب شده جهت بیان و کلونینگ، وکتور pET32a انتخاب گردید. بررسی و ایجاد جایگاههای برش آنزیم محدودکننده توسط نرمافزار NEBcutter و در نهایت ساخت توالی ژن طراحیشده توسط GenScript (آلمان) درون وکتور pET32a انجام شد(24).
آمادهسازی و تراریختی میزبان کلونینگ:
سلولهایTOP10 جهت پذیرش وکتور با روش شیمیایی با استفاده از کلریدکلسیم مستعدسازی گشته و تراریختی و انتقال وکتورpET32a-LZ/SCF به درون سلولهای مستعد به منظور افزایش تعداد وکتور با استفاده از روش شوک حرارتی انجام شد.
استخراج وکتور نوترکیب و بررسی صحت همسانهسازی ژن هدف در وکتور:
پس از ایجاد کلنیهای اختصاصی، وکتور نوترکیب pET32a-LZ/SCF از باکتری TOP10 استخراج شد. بررسی الگوی هضم آنزیمی با استفاده از آنزیمهای محدودکنندهNcoI و XhoIجهت بررسی خلوص وکتور و صحت همسانهسازی ژن هدف در وکتور pET32a انجام شد. الکتروفورز ژل آگارز جهت بررسی نتیجه انجام گرفت.
بیان پروتئین نوترکیب LZ/SCF :
پس از آمادهسازی سلولهای باکتری اریگامی جهت پذیرش وکتور و تراریختی با وکتورpET32a-LZ/SCF ، پروتئین نوترکیب LZ/SCF در باکتری اریگامی بیان گردید. بررسی بیان پروتئین نوترکیب توسط روش SDS–PAGE صورت گرفت. برای تأیید هویت پروتئین نوترکیب، وسترنبلات انجام گرفت.
یافتهها
طراحی و پیشبینی ساختار اول، دوم و سوم پروتئین نوترکیب:
در گام نخست توالی آمینواسیدی فاکتـور سلـول بنیادی
(SCF) و توالـی زیـپلوسیـن از بانـک اطلاعـات پروتئین
Uniprotkb به دست آمد. از توالی SCF(165) برای طراحی پروتئین نوترکیب استفاده شد که پیوندهای دیسولفیدی لازم برای عملکرد صحیح پروتئین و نقاط اتصال به گیرنده SCF (c-Kit) نیز در این محدوده توالی وجود دارد. پس از بررسی ساختار توالیهای زیپلوسین در اتصال با پروتئین SCF، توالی زیپلوسین مربوط به پروتئین Myocilin انتخاب گردید.
در طراحی ساختمان اول پروتئین نوترکیب، زیپلوسین برگرفته از پروتئین میوسیلین، Nترمینال و توالی مربوط به SCF، Cترمینال پروتئین طراحیشده را تشکیل میدهد. ساختار دوم پروتئین نوترکیب توسط نرمافزار PSIpred بررسی گشته و نتایج مدلسازی همولوژیکی حاصل از برنامه SWISS-MODEL نشاندهنده ساختاری از پروتئین نوترکیب LZ/SCF میباشد که در آن نحوه اتصال توالی پروتئین Leucine Zipper و SCF و ساختمان سهبعدی پروتئیـن نوترکیـب بـه صـورت مطلوب میباشد(شکل 1).

نتایج به دست آمده از پیشبینی ساختار سوم پروتئین نوترکیب LZ/SCF توسط نرمافزار Chimera به وسیله نرمافزارPyMOL مشاهده شد. مطابق انتظار در پروتئین نوترکیب، دومین تشکیلدهنده SCF واجد چهار هلیکس و دو beta-sheet و زیپلوسین نیز متشکل از یک هلیکس خطی میباشد(شکل 2).

بررسی پایداری ساختار:
پایداری ساختار سوم پیشبینی شده برای پروتئین نوترکیب با استفاده از نمودار راماچاندران، نشاندهنده قرارگیری 8/95% از ریشههای آمینواسیدی در ناحیه مطلوب و 8/2% در ناحیه مجاز است در نتیجه پروتئین نوترکیب LZ/SCF از پایداری مناسبی در محیط سلول برخوردار میباشد(شکل 3).

بهینهسازی توالی نوکلئوتیدی:
به منظور بهینهسازی توالی پروتئین نوترکیب طراحی شده جهت بیان در میزبان پروکاریوتی(E. coli K12) از نرمافزار آنلاین Optimizer استفاده شد.
با توجه به نتایج حاصل از نرمافزار NEBcutter، آنزیمهای محدودکننده NcoI و XhoIانتخاب شدند. توالی محل اثر برش این آنزیمها به سکانس نوکلئوتیدی طراحی شده اضافه گشت. در نهایت توالی ژنی ساخته شده (GenScript، آلمان) و به درون وکتور pET32a وارد شد.
آمادهسازی و تراریختی میزبان کلونینگ:
پس از آمادهسازی سلولهای باکتری TOP10 (انستیتو پاستور، ایران) جهت پذیرش وکتور و نیز تراریختی این باکتریها با وکتور نوترکیب، باکتریها در محیط کشت انتخابی LB agar (Luria Bertani Broth) جامد حاوی آنتیبیوتیک آمپیسیلین (ژن مقاومت به آمپیسیلین بر روی وکتور pET32a-LZ/SCF وجود دارد) کشت و به صورت کلنی ظاهر شدند. این امر نشاندهنده موفقیت در تراریختی باکتریهای TOP10 با وکتور نوترکیب pET32a-LZ/SCF و ورود آن به ژنوم باکتری میباشد.
استخراج وکتور نوترکیب و بررسی صحت همسانهسازی ژن هدف در وکتور:
استخراج وکتور نوترکیب pET32a-LZ/SCF از باکتریهای E.coli سویه TOP10 با استفاده از کیت استخراج وکتور(Favorgene ، تایوان) به روش Miniprep انجام شد. جهت بررسی الگوی هضم آنزیمی، وکتور نوترکیب با استفاده از آنزیمهای محدودکننده NcoI و XhoI در بافر مشترک(Tango 2X) شکسته شد. بررسی کیفیت وکتور استخراج شده با استفاده از الکتروفورز ژل آگارز 1 درصد انجام شد. بررسی اندازه قطعات حاصل از هضم آنزیمی و حضور قطعه ژنی با اندازه مورد نظر، صحت همسانهسازی را تأیید نمود(نمودار 1).
آمادهسازی و تراریختی میزبان
|
659 bp
|
|
659 bp
|
 بیانی:
بیانی:مستعدسازی سلولهای باکتری اریگامی(انستیتو پاستور، ایران) جهت پذیرش وکتور با استفاده از کلریدکلسیم و تراریختی و انتقال وکتورpET32a-LZ/SCF به داخل سلولهای مستعد شده توسط شوک حرارتی انجام شد. سپس باکتریها در محیط کشت انتخابی LB agar جامد حاوی آنتیبیوتیک آمپیسیلین و آنتیبیوتیکهای کانامایسین و تتراسایکلین (ژنهای مقاوم به این آنتیبیوتیکها در باکتری اریگامی وجود دارد) رشد کرده و کلنی تشکیل دادند که نشاندهنده موفقیت در تراریختی باکتریهای اریگامی با وکتور نوترکیب pET32a- LZ/SCF
و ورود آن به ژنوم باکتری است.

بیان پروتئین نوترکیب LZ/SCF :
جهت بیان پروتئین نوترکیب LZ/SCFپس از گرماگذاری کشتها به مدت یک شب در انکوباتور، از آن کشت تازه تهیه شد و القای بیان پروتئین نوترکیب LZ/SCF توسط IPTG (سیگما، آمریکا) انجام شد. بررسی بیان فیوژن پروتئین نوترکیب LZ/SCFدر اریگامی، توسط الکتروفورز پروتئینها با استفاده از روش SDS-PAGE انجام شد. نتایج حاصل از الکتروفورز(قسمت الف نمودار 2) نشاندهنده بیان پروتئین نوترکیب LZ/SCFدر ستون 2 پس از گذشت 6 ساعت پس از القا توسط IPTG در مقایسه با نمونههای قبل از القا در ستون 3 میباشد. در این میان در بررسی فواصل زمانی 2، 4 و 6 ساعت پس از القا با یکدیگر به نظر میرسد که باکتری در زمان 6 ساعت پس از القا توسط IPTG ، بیشترین میزان بیان پروتئین نوترکیب LZ/SCF را نشان داده و زمان مناسبتری برای برداشت پروتئین است(قسمت ب نمودار 2).
از میان کلنیهای تراریخت شده باکتری اریگامی با وکتور نوترکیب PET32a-LZ/SCF ، چهار کلنی مجزا و کوچک انتخاب شده و جهت بررسی بهترین غلظــت IPTG جهـت القـای بیان باکتری، از غلظتهای متفاوت IPTG 1/0- 5/0- 1- 2 میلیمولار استفاده شد که غلظت 1 میلیمولار بهترین میزان غلظت برای القای باکتری جهت بیان پروتئین نوترکیب LZ/SCF میباشد(قسمت ج نمودار 2).


تأیید هویت پروتئین نوترکیب LZ/SCF :
در انتها به دلیل این که توالی پروتئین نوترکیب LZ/SCF دارای His-Tag میباشد، پس از بیان هویت آن توسط وسترنبلات با استفاده از آنتیبادیهای مونوکلونال Anti His-Tag متصل به آنزیم HRP تأیید شد(نمودار 3).
بحث
نظـر به اهمیت حضور SCF در بقا، تکثیر و تمایز انواع
مختلـف سلـــولهـای بنیـادی خونسـاز، پیـشســازهای
ملانوسیتها، ماستسلها و سلولهای زایا و در راستای پیشرفت روزافزون بیوتکنولوژی و ژندرمانی در دهههای اخیر، SCF به عنوان یکی از گزینههای مهم در فعالیتهای بیوتکنولوژی شناخته شده است. SCF به نحو گستردهای در مطالعههای سلولهای بنیادی، بررسی روند مولکولی سرطانها و مهاجرت سلولهای سرطانی و مطالعههای مولکولی در انواع کمخونیها و حتی ایجاد روشهای درمانی جدید مورد استفاده است. بدین منظور در بسیاری از مطالعههای موجود در این حیطه، تولید SCF به شیوه نوترکیب مورد توجه قرار گرفته است. اما در این مسیر، صرف دستیابی به SCFنوترکیب اغناکننده و به صرفه نمیباشد؛ بلکه تولید SCF نوترکیبی که واجد ساختاری پایدارتر و عملکردی بیشتر از فرم طبیعی باشد، مطلوبتر خواهد بود.
در طی مطالعهای در سال 1997 مقایسه دو فرم دایمر کووالان (فرم نوترکیب) و غیرکووالان (فرم طبیعی) SCF نشاندهنده افزایش در تعداد کلنیهای مشاهده شده از SCF نوترکیب و توانایی تحریک رشد سلولهای مشتق از میزبانهای مورد نظر مطالعه و نیز پایداری بالاتر در مقایسه با SCF طبیعی بود(5). با توجه به این مطالعه که سعی بر ارتقای SCF از طریق تأثیر بر نحوه دایمریزاسیون مونومرهای SCF شده و باعث به دست آمدن راندمان بالاتری در بیان و پایداری SCF گشته است؛ ایده ارتقای SCF توسط تقویت و یا تغییر نحوه دایمریزاسیون SCF شکل گرفت. در مطالعه عنوان شده با تداخل در ساختار فضایی پروتئین به نتیجه موردنظر دست یافتند؛ در نتیجه با توجه به این که زیپلوسین یکی از مواردی است که به صورت طبیعی قادر به دایمریزاسیون توالیهای متصل به خود میباشد و با توجه به مطالعههای بسیاری که در این زمینه انجام شده است و نظر به تأثیرات زیپلوسین در پایداری و افزایش فعالیت دومین کاتالیتیک، توالیهای پروتئینهایی که موتیف زیپلوسین به صورت اتصال طبیعی یا نوترکیب به آنها متصل گشته است(9-7). در مطالعه پیشرو با اضافه کردن موتیف زیپلوسین به توالی SCF ، در واقع از زیپلوسین به عنوان ابزاری طبیعی جهت دستیابی به نتایج موردنظر بدون تداخل در ساختمان فضایی پروتئین استفاده شد. در مطالعه انجام شده در سال 2005، دایمریزاسیون مونومرهای پروتئین توسط زیپلوسین موجود در ساختار آن نشاندهنده تأثیر زیپلوسین بر پایداری این پروتئین در انواع شرایط سخت بافری میباشد(7). در پژوهش صورت گرفته در سال 2000 نیز جهت بررسی اتصال زیپلوسین به صورت نوترکیب به پروتئینها، در in vivo و in vitro ، افزایش فعالیت دومین کاتالیتیک متصل به زیپلوسین (فرم نوترکیب) نسبت به دومین کاتالیتیک (فرم طبیعی) مشاهده گردید(9).
تاکنون مطالعههای صورت گرفته برSCF در ایران، حول محور کلون و بیان توالی SCF بوده است. در مطالعهای در سال 2014 در ایران، توالی کدکننده فرم محلول SCF را کلون نمودند اما در مورد بیان آن گزارشی ارائه نگردید(25). همچنین در مطالعهای در سال 2017 در جهت بررسی بیان SCF در ایران، توالی کدکننده مونومر SCF کلون و در سویه Rosetta-gami بیان شد(4).
هدف مطالعه حاضر با توجه به مطالعههای عنوان شده، طراحی و بیان توالی پروتئینی نوترکیب LZ/SCF بود. جهت تحقق هدف پیشرو پس از بررسی پایگاههای اطلاعاتی مختلف، توالی آمینواسیدی فاکتور سلول بنیادی (SCF) و توالی زیپلوسین مربوط به پروتئین میوسیلین به دست آمد. جهت بررسی ساختار دوم پروتئین نوترکیب از نرمافزار PSIpred استفاده شد. ساختمان سه بعدی پروتئین نوترکیب LZ/SCF توسط برنامه Swiss-model بررسی و در نتایج مدلسازی همولوژیکی، نحوه اتصال توالی پروتئین Leucine Zipper و SCF به هم و ساختمان سه بعدی آن به صورت مطلوب میباشد. بررسی ساختار سوم توالیهای انتخابی نیز تحت چیدمانهای متفاوت توسط نرمافزار Chimera انجام گرفت. نتایج به دست آمده از پیشبینی ساختار سوم پروتئین نوترکیب LZ/SCF توسط نرمافزار Chimera نشاندهنده فولد مناسب و تشکیل ساختار کامل سازه طراحی شده میباشد. پایداری ساختار سوم پروتئین نوترکیب LZ/SCF با استفاده از نمودار راماچاندران بررسی شـد کـه نشاندهنـده پایـداری مناســب پروتئین نوترکیب
LZ/SCF در محیط سلول است.
از آن جا که در مطالعه صورت گرفته در سال 2017 جهت بیان کدونهای نادر، از سویه Rosetta-gami به عنوان میزبان بیانی استفاده شد(4). در مطالعه حاضر به دلیل استفاده از سویه اریگامی به عنوان میزبان بیانی و بهینهسازی توالی برای میزبان انتخاب شده توسط نرمافزار آنلاین اپتیمایزر، تمامی کدونها در اریگامی بیان خواهند شد. با توجه به نتایج حاصل از نرمافزار NEBcutter، آنزیمهای محدودکننده NcoI و XhoIانتخاب شدند. توالی محل اثر برش این آنزیمها که برای بیان پروتئین در میزبان پروکاریوتی ضروری است، به سکانس نوکلئوتیدی طراحی شده اضافه گشت. در نهایت توالی ژنی توسط(GenScript، آلمان) سنتز و درون وکتور pET32a سابکلون گردید.
جهت همسانهسازی سازه ژنی از باکتری TOP10 (انستیتو پاستور، ایران) به عنوان میزبان کلونینگ استفاده شد. استخراج وکتور pET32a- LZ/SCF از میزبان کلونینگ با استفاده از کیت استخراج پلاسمید(Favorgene ، تایوان) به روش Miniprep انجام شد. به منظور مشاهده و اطمینان از صحت و خلوص وکتور استخراج شده، محصول واکنش هضم آنزیمی، با استفاده از الکتروفورز در ژل آگارز مورد بررسی قرار گرفت.
در مطالعههای پیشین پس از بیان پروتئین نوترکیب در باکتری E.coli ، نیاز به Refolding پروتئین نوترکیب جهت تشکیل پیوندهای دیسولفیدی بود(26). از آن جا که دو پیوند دیسولفیدی SCF جهت اتصال و فعالیت زیستی گیرنده ضروری است و محیط باکتری E.coli به دلیل احیاکننده بودن سبب شکست پیوندهای دیسولفیدی موجود در پروتئین نوترکیب LZ/SCF میگردد، در نتیجه در مطالعه حاضر از باکتری اریگامی استفاده شد که موتانت برگرفته از سویه E.coli K12 میباشد زیرا سویه مهندسیشده اریگامی با دارا بودن جهشهایی در ژنهای تیوردکسینردوکتاز و گلوتاتیونردوکتاز، امکان تشکیل باندهای دیسولفیدی ضروری در ساختار پروتئین را فراهم میآورد. در مطالعه انجام گرفته در سال 2015، جهت بیان پیوندهای دیسولفیدی، توالی تیوردوکسینردوکتاز را با وکتور، بیان همزمان(coexpression) نموده که در مطالعه
حاضر مرتفع نمودن این نیاز با اضافه نمودن توالی تیوردوکسینردوکتاز به توالی پروتئین نوترکیب طراحی شده انجام گرفت(27). لازم به ذکر است که SCF در سلولهای پستانداران به صورت N-glycosylate است با این وجود ثابت شده است که حضور یا عدم حضور کربوهیدرات در SCF برای فعالیت زیستی ضروری به نظر نمیرسد. به همین دلیل بیان پروتئین نوترکیب LZ/SCF در باکتری E. coli باعث اختلال در فعالیت زیستی آن نخواهد شد. در نهایت بیان فیوژن پروتئین LZ/SCF در میزبان بیانی اریگامی به روش SDS – PAGE بررسی شد و سپس با روش وسترنبلات هویت پروتئین تأیید گشت.
نتیجهگیری
نتایج حاصل از مطالعه حاضر، تأیید ساختار مناسب سازه طراحی شده توسط نرمافزارهای مورد استفاده و موفق بودن کلون وکتور نوترکیب pET32a-LZ/SCF در میزبان باکتریایی کلونینگ TOP10)) و حصول کلونهای اختصاصی میباشد. بیان پروتئین LZ/SCF در میزبان باکتریایی بیان اریگامی نیز با موفقیت انجام شد.
تشکر و قدردانی
این مقاله حاصل پایاننامه دانشجویی دوره کارشناسی ارشد رشته بیوتکنولوژی پزشکی مرکز تحقیقات مؤسسه عالی آموزشی و پژوهشی طب انتقال خون میباشد. بودجه این تحقیق توسط مؤسسه عالی آموزشی و پژوهشی طب انتقال خون تأمین گردیده و همه مراحل علمی پروژه در مرکز تحقیقات سازمان انتقال خون ایران انجام شده است. پروپوزال این پایاننامه با دریافت کد اخلاق IR.TMI.REC.1397.002 در کمیته تخصصی اخلاق در پژوهشهای زیست پزشکی تصویب شده است.
نوع مطالعه: پژوهشي |
موضوع مقاله:
بيوتكنولوژي
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |






